30 días de gráficos en R
24 May 2020 161 minsIntroducción
A raíz de que el 12 de mayo se conmemora el nacimiento de Florence Nightingale, la enfermera creadora del diagrama de área polar y referente femenina de la visualización de datos, la comunidad @R4DS_es lanzó un lindo desafío, proyecto, uno distinto por día. Esta es la lista completa:
| día | fecha | desafío | Completado? |
|---|---|---|---|
| 1 | 12 de mayo | barras / columnas | ✓ |
| 2 | 13 de mayo | líneas | ✓ |
| 3 | 14 de mayo | puntos / burbujas | ✓ |
| 4 | 15 de mayo | gráficos con facetas | ✓ |
| 5 | 16 de mayo | diagramas de arco | ✓ |
| 6 | 17 de mayo | gráficos de donut | ✓ |
| 7 | 18 de mayo | gráficos ridgeline | ✓ |
| 8 | 19 de mayo | gráficos de contorno | ✓ |
| 9 | 20 de mayo | gráficos de áreas apiladas | ✓ |
| 10 | 21 de mayo | ¡explorar paletas de colores! | ✓ |
| 11 | 22 de mayo | mapas de calor (heatmap) | ✓ |
| 12 | 23 de mayo | gráficos de paleta (lollipop) | ✓ |
| 13 | 24 de mayo | visualizar datos temporales | ✓ |
| 14 | 25 de mayo | gráficos de rectángulos/árbol | ✓ |
| 15 | 26 de mayo | dendorgamas | ✓ |
| 16 | 27 de mayo | gráficos de waffle | ✓ |
| 17 | 28 de mayo | diagramas de sankey | ✓ |
| 18 | 29 de mayo | visualizar datos espaciales | ✓ |
| 19 | 30 de mayo | gráficos de flujo (stream graph) | ✓ |
| 20 | 31 de mayo | redes | ✓ |
| 21 | 1 de junio | gráficos con anotaciones | ✓ |
| 22 | 2 de junio | visualizar datos textuales | ✓ |
| 23 | 3 de junio | gráficos de proyección solar (sunburst) | ✓ |
| 24 | 4 de junio | coropletas | ✓ |
| 25 | 5 de junio | gráficos de violín | ✓ |
| 26 | 6 de junio | diagramas de marimekko | ✓ |
| 27 | 7 de junio | ¡gráficos animados! | ✓ |
| 28 | 8 de junio | diagramas de cuerdas | ✓ |
| 29 | 9 de junio | gráficos de coordenadas paralelas | ✓ |
| 30 | 10 de junio | diagramas de área polar o de Florence Nightingale | ✓ |
Objetivos
En lo personal, mi idea es aprovechar este desafío para:
- Profundizar el conocimiento de Ggplot2, ya que intentaremos resolver todos usando este paquete
- Probar y eventualmente ajustar, mi tema personalizado
ggelegant - Tratar de usar datos actuales y no apoyarme en gráficas de ejemplo ya resueltas
Día 1: Gráfico de barras / Columnas
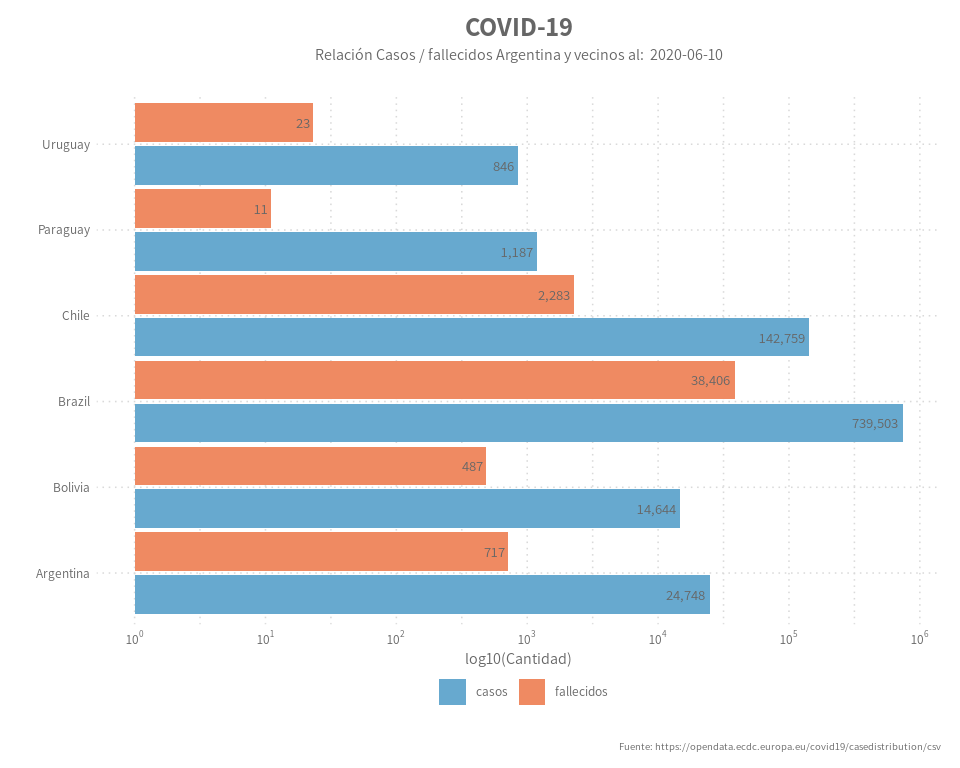
Una gráfica de barras o columnas, tal vez una de las más clásicas, muestra comparaciones numéricas entre una variable discreta y los valores continuos que toma cada una de estas variables o “categorías”. A diferencia de un histograma, los gráficos de barra no muestran un desarrollo continuo entre las categorías.
Este ejemplo muestra las diferencia de situación con respecto al COVID-19 en Argentina y los países vecinos, en cuanto a cantidad de casos y fallecidos. Usamos una escala logarítmica en el caso del eje y para que los enormes números de Brasil no nos oculten los datos del resto.
Preparamos los datos:
library("tidyverse")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM",
# stringsAsFactors = FALSE)
# Datos para reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
last_date <- max(as.Date(covid.data$dateRep,"%d/%m/%Y"))
covid.data %>%
filter(countriesAndTerritories %in% c('Argentina','Brazil', 'Chile', 'Bolivia', 'Paraguay', 'Uruguay')) %>%
group_by(countriesAndTerritories) %>%
summarize(casos = sum(cases), fallecidos = sum(deaths)) %>%
ungroup() %>%
select(pais = countriesAndTerritories, casos, fallecidos) %>%
gather(referencia, cantidad, -pais) -> plot_data
kable(plot_data)
| pais | referencia | cantidad |
|---|---|---|
| Argentina | casos | 24748 |
| Bolivia | casos | 14644 |
| Brazil | casos | 739503 |
| Chile | casos | 142759 |
| Paraguay | casos | 1187 |
| Uruguay | casos | 846 |
| Argentina | fallecidos | 717 |
| Bolivia | fallecidos | 487 |
| Brazil | fallecidos | 38406 |
| Chile | fallecidos | 2283 |
| Paraguay | fallecidos | 11 |
| Uruguay | fallecidos | 23 |
La gráfica
plot_data %>%
ggplot(aes(x=pais, fill=referencia, y=cantidad)) +
geom_col(position=position_dodge(width=1)) +
geom_text(aes(label = format(cantidad, digits=0, big.mark = ',')), vjust = .6, hjust=1.1,
position = position_dodge(width=1)) +
coord_flip() +
scale_y_log10(
breaks = scales::trans_breaks("log10", function(x) 10^x),
labels = scales::trans_format("log10", scales::math_format(10^.x))
) +
labs(title = paste("COVID-19"),
subtitle = paste("Relación Casos / fallecidos Argentina y vecinos al: ", last_date) ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "log10(Cantidad)",
x = ""
) +
scale_fill_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Assistant") +
theme(plot.caption=element_text( margin=margin(1, 0, -.1, 0, "cm")),
plot.subtitle = element_text(margin=margin(0, 0, .8, 0, "cm")))
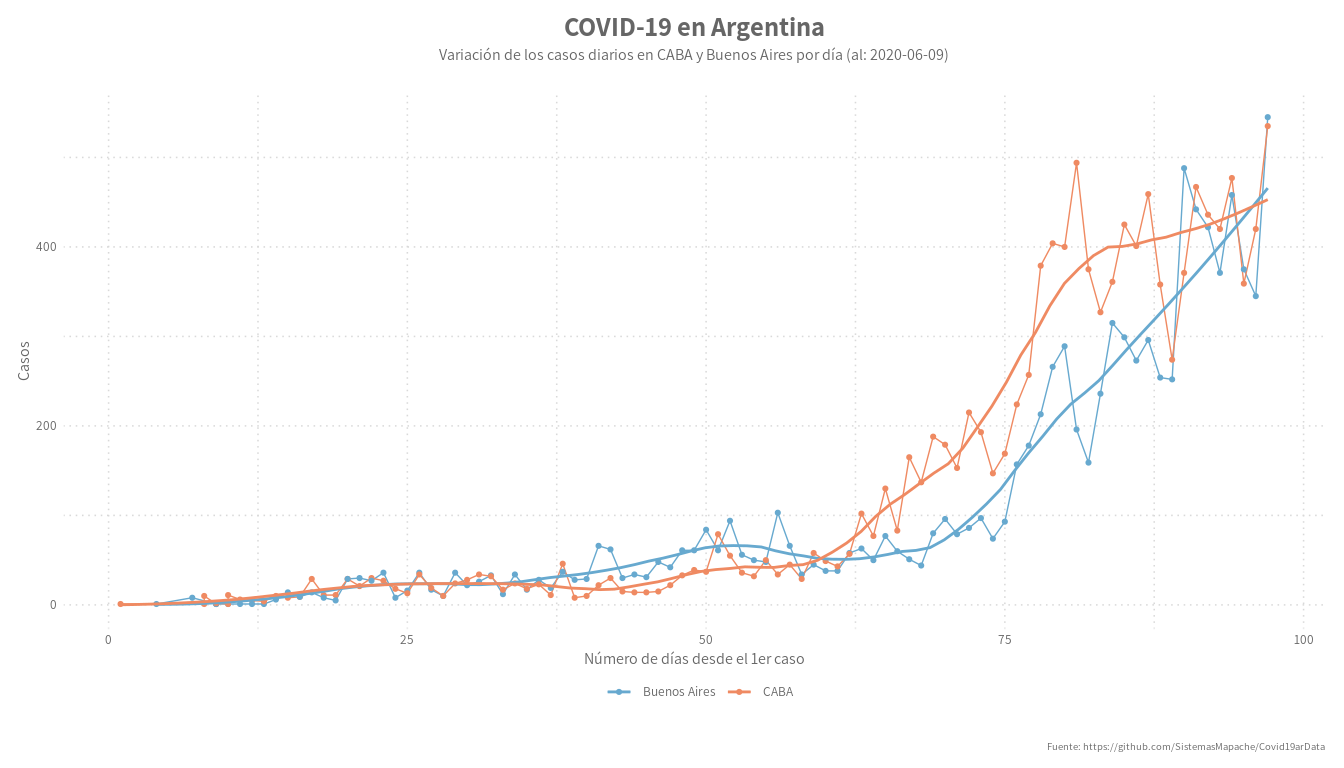
Día 2: Gráfico de líneas
Son gráficos que muestras la evolución de una o más variables continuas en un determinado intervalo, generalmente de tiempo. Lo habitual es que el eje Y tenga un valor cuantitativo y el eje X tiene la secuencia que representa el intervalo, En el plano cartesiano, se establecen los puntos X, Y y luego se van conectando estos mediante líneas rectas.
En este ejemplo, la idea es mostrar la evolución de los casos de COVID-19 en las dos provincias más importantes en cantidad de casos de la Argentina, desde el primer infectado (día 1), el eje x muestra el número de días y el y la cantidad de casos detectados dicho día. Dibujamos puntos en cada x, y y luego unimos cada uno con un segmento, adicionalmente agregamos una curva que refuerza la visión de la tendencia.
Los datos
library("tidyverse")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
covid.data %>%
filter(osm_admin_level_4 %in% c('CABA', 'Buenos Aires')) %>%
mutate(fecha = as.Date(fecha, "%d/%m/%Y")) %>%
select(dia=dia_inicio, distrito=osm_admin_level_4, cantidad=nue_casosconf_diff) -> plot_data
kable(head(plot_data))
| dia | distrito | cantidad |
|---|---|---|
| 1 | CABA | 1 |
| 4 | Buenos Aires | 1 |
| 7 | Buenos Aires | 8 |
| 8 | CABA | 1 |
| 9 | Buenos Aires | 1 |
| 9 | CABA | 1 |
La gráfica
plot_data %>%
ggplot(mapping=aes(x=dia, color=distrito, y=cantidad)) +
geom_line() +
geom_point() +
geom_smooth(method = 'loess',
formula = 'y ~ x', alpha = 0.2, size = 1, span = .3, se=FALSE) +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Variación de los casos diarios en CABA y Buenos Aires por día (al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Casos",
x = "Número de días desde el 1er caso"
) +
scale_color_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Assistant")
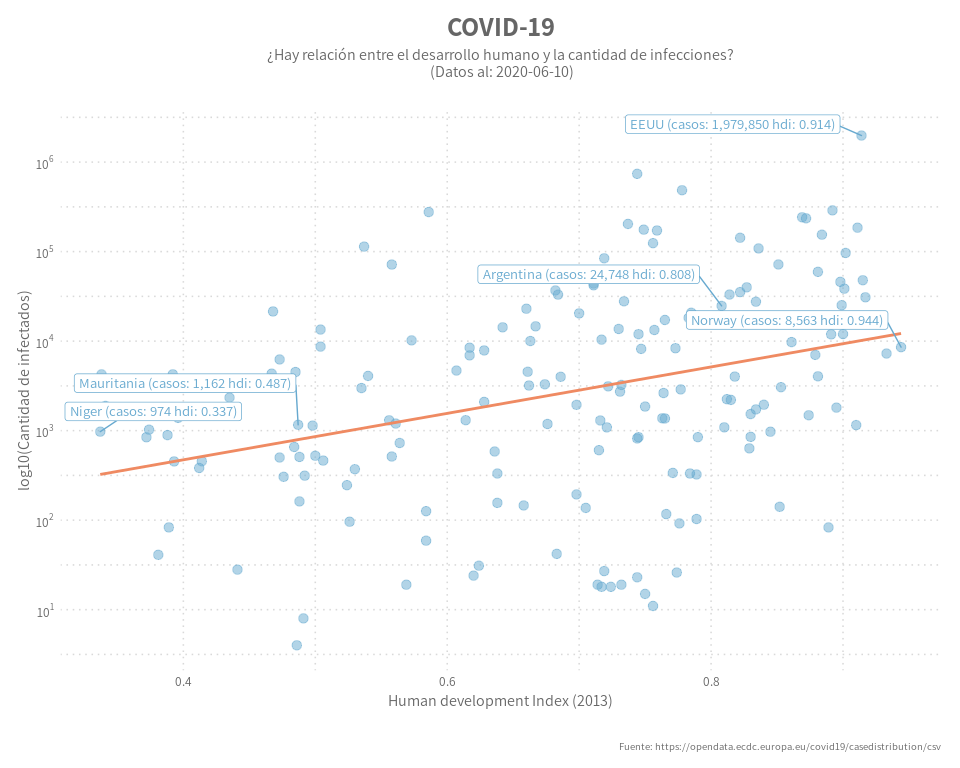
Día 3: Una gráfica de puntos
También se los conoce como gráficos de dispersión o “Scatterplot”. Suelen intentar mostrar la relación entre dos variables continuas por medio de los patrones que se forman.
En este ejemplo, me pregunto sí, ¿Hay relación entre el desarrollo humano y la cantidad de infecciones?, para esto armamos un gráfico de dispersión dónde cada país tiene un punto que correlaciona las variables: números de infectados (x) y el índice de desarrollo humano (y), además agregamos unas etiquetas que marcan los países en el extremo de cada variable y la Argentina y una recta de regresión a modo de tendencia.
Los datos
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Para descarga de los datos actualizados
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# hdi <- read.csv("https://data.humdata.org/dataset/05b5d8f1-9e7f-4379-9958-125c203d12ac/resource/4a7fd374-7e35-4c04-b7c8-25e5943aa476/downlo
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
hdi <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/hdi.Rda","rb"))
hdi %>%
group_by(country_code) %>%
arrange(year) %>%
slice(n()) %>%
select(country_code, country, year, value) -> last_hdi
last_date <- max(as.Date(covid.data$dateRep,"%d/%m/%Y"))
paises_de_interes <- c( 'Argentina',
"Niger", "Norway", "EEUU", "Mauritania")
covid.data %>%
group_by(countriesAndTerritories, countryterritoryCode) %>%
summarize(casos = sum(cases), fallecidos = sum(deaths)) %>%
ungroup() %>%
inner_join(last_hdi,
by = c("countryterritoryCode" = "country_code")
) %>%
mutate(pais = ifelse(countriesAndTerritories == 'United_States_of_America', 'EEUU', countriesAndTerritories)) %>%
select(pais, casos, HDI = value) %>%
mutate(pais_etiquetado = ifelse(pais %in% paises_de_interes, paste0(pais, " (casos: ", format(casos, digits=0, big.mark = ',', trim=TRUE), " hdi: ", HDI, ")"), NA)) -> plot_data
kable(head(plot_data))
| pais | casos | HDI | pais_etiquetado |
|---|---|---|---|
| Afghanistan | 27532 | 0.468 | NA |
| Albania | 1788 | 0.716 | NA |
| Algeria | 11385 | 0.717 | NA |
| Andorra | 855 | 0.830 | NA |
| Angola | 155 | 0.526 | NA |
| Antigua_and_Barbuda | 26 | 0.774 | NA |
La gráfica
plot_data %>%
ggplot(aes(x=HDI, y=casos)) +
geom_point(color = "#67a9cf", alpha=.5, size=3) +
geom_smooth(method = 'lm',formula='y ~ x', se=FALSE, color="#ef8a62") +
geom_label_repel(mapping = aes(label = pais_etiquetado),
color="#67a9cf",family = "Assistant", vjust = -1.2, hjust = 1.1) +
# nudge_x = 1, nudge_y = 5, color="#67a9cf",
# vjust = -2, family = "Assistant",
# direction = "y",
# hjust = 2) +
scale_y_log10(
breaks = scales::trans_breaks("log10", function(x) 10^x),
labels = scales::trans_format("log10", scales::math_format(10^.x))
) +
labs(title = paste("COVID-19"),
subtitle = paste0("¿Hay relación entre el desarrollo humano y la cantidad de infecciones?\n (Datos al: ", last_date, ")") ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "log10(Cantidad de infectados)",
x = "Human development Index (2013)"
) +
theme_elegante_std(base_family = "Assistant")
Día 4: Facetas
Las “Facetas” no es un tipo de gráfico sino más bien una forma de mostrar uno de estos. Cuando se tiene múltiples variables, muchas veces resulta confuso verlas todas en un mismo gráfico, el facetado permite dividir cada variable o grupo en múltiples graficos similares.
Los datos
library("tidyverse")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
covid.data %>%
mutate(fecha = as.Date(fecha, "%d/%m/%Y")) %>%
select(dia=dia_inicio, distrito=osm_admin_level_4, cantidad=nue_casosconf_diff) %>%
complete(distrito, dia, fill=list(cantidad=0)) %>%
arrange(distrito, dia) -> data
data %>%
inner_join(data %>%
group_by(distrito) %>%
summarize(cantidad = sum(cantidad)) %>%
arrange(-cantidad) %>%
top_n(9), by = c("distrito"), suffix=c("",".y")) %>%
select(dia, distrito, cantidad) %>%
arrange(dia, distrito) -> plot_data
kable(head(plot_data))
| dia | distrito | cantidad |
|---|---|---|
| 1 | Buenos Aires | 0 |
| 1 | CABA | 1 |
| 1 | Chaco | 0 |
| 1 | Córdoba | 0 |
| 1 | Mendoza | 0 |
| 1 | Neuquén | 0 |
La gráfica
plot_data %>%
ggplot(mapping=aes(x=dia, y=cantidad)) +
geom_line(color="#67a9cf") +
geom_point(color="#67a9cf") +
geom_smooth(method = 'loess',
formula = 'y ~ x', alpha = 0.2, size = 1, span = .3, se=FALSE, color="#ef8a62") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Variación de casos diarios en los distritos con más casos (al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Número de casos",
x = "Número de días desde el 1er caso"
) +
facet_wrap(~distrito,scales="free") +
theme_elegante_std(base_family = "Assistant")
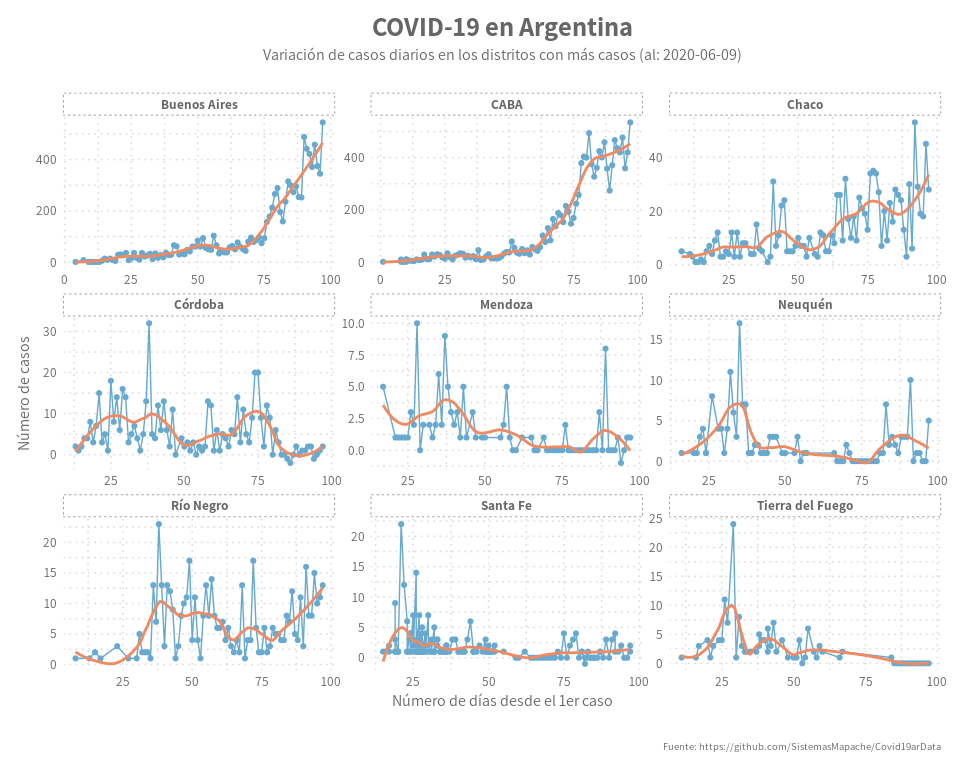
Una linda visualización mediante geofaceteAR:
Este paquete permite facetar siguiendo la estructura geografica, en esta caso el de las provincias de Argentina, da una visión muy atractiva y clara de los datos, ya no es necesario iterar visualmente hasta encontrar la información de un estado, sino que accedemos a la gráfica de forma más directa.
library("geofaceteAR")
argentina_grid <- data.frame(
col = c(1, 3, 5, 1, 2, 1, 3, 4, 2, 2, 4, 1, 3, 3, 4, 1, 2, 2, 1, 1, 2, 1, 1, 1),
row = c(1, 2, 2, 2, 2, 3, 3, 3, 3, 4, 4, 4, 4, 5, 5, 5, 5, 6, 6, 7, 7, 8, 9, 10),
code = c("AR-Y", "AR-P", "AR-N", "AR-A", "AR-T", "AR-K", "AR-H", "AR-W", "AR-G", "AR-X", "AR-E", "AR-F", "AR-S", "AR-B", "AR-C", "AR-J", "AR-D", "AR-L", "AR-M", "AR-Q", "AR-R", "AR-U", "AR-Z", "AR-V"),
name_es = c("Jujuy", "Formosa", "Misiones", "Salta", "Tucumán", "Catamarca", "Chaco", "Corrientes", "Santiago del Estero", "Córdoba",
"Entre Ríos", "La Rioja", "Santa Fe", "Buenos Aires", "CABA", "San Juan", "San Luis", "La Pampa", "Mendoza", "Neuquén", "Río Negro",
"Chubut", "Santa Cruz", "Tierra del Fuego"),
stringsAsFactors = FALSE
)
data %>%
filter(distrito!='Indeterminado') %>%
ggplot(mapping=aes(x=dia, y=cantidad)) +
geom_line(color="#67a9cf") +
geom_point(color="#67a9cf") +
geom_smooth(method = 'loess',
formula = 'y ~ x', alpha = 0.2, size = 1, span = .3, se=FALSE, color="#ef8a62") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Variación de casos diarios en los distritos con más casos (al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Número de casos",
x = "Número de días desde el 1er caso"
) +
facet_geo(~ distrito, grid = argentina_grid, scales = "free_y") +
theme_elegante_std(base_family = "Assistant")
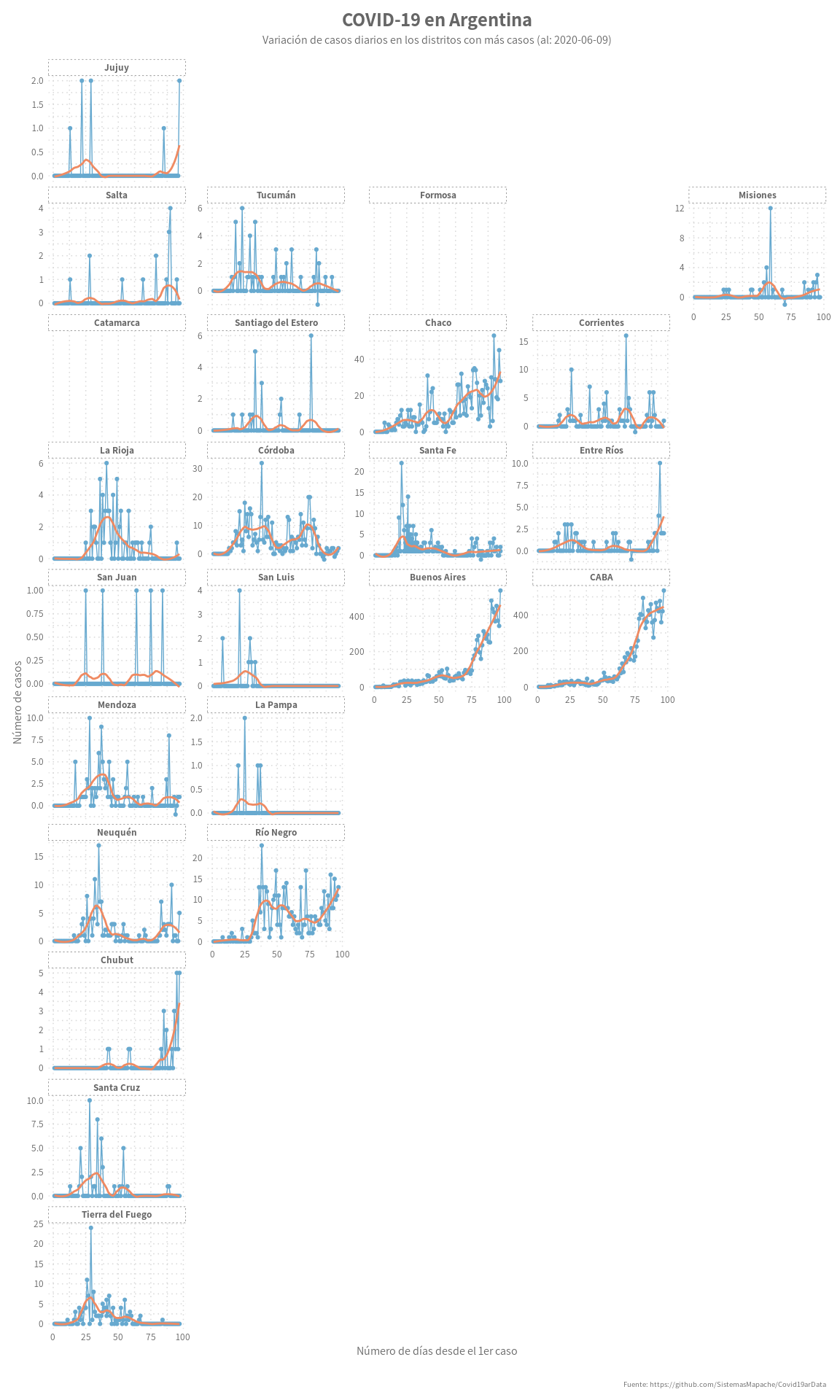
Día 5: Un gráfico de Arcos
Los gráficos de arco forman parte de los diagramas de red, en este caso los nodos se colocan sobre un eje horizontal y la líneas o arcos señalan las relaciones entre cada nodo. Eventualmente se puede ajustar el grosor de la líne para incluir una nueva dimensión. En lo personal me resulta difíciles de leer pero entiendo que para algunas aplicaciones específicas pueden ser útiles. En este ejemplo, trato de mostrar las relaciones musicales interpresonales de Luis Alberto Spinetta en su primera etapa como músico.
Los datos
library("tidyverse")
library("tidygraph")
library("ggraph")
library("igraph")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
data <- structure(list(persona = structure(c(10L, 10L, 10L, 10L, 10L,
10L, 6L, 6L, 12L, 12L, 12L, 12L, 8L, 9L, 2L, 7L, 7L, 3L, 3L,
1L, 11L, 5L, 4L, 4L, 13L), .Label = c("Angel del Guercio", "Carlos Xartuch",
"Daniel Albertelli", "Edelmiro Molinari", "Eduardo Miró", "Emilio del Guercio",
"Guido Meda", "Hector Nuñez", "Horacio Soria", "Luis Alberto Spinetta",
"Ricardo Miró", "Rodolfo Garcia", "Santiago Novoa"), class = "factor"),
grupo = c(" Bundleman", " Los Larkings", " Los Masters",
" Los Mods", " Los Sbirros", " Almendra (1967-1969)", " Bundleman",
" Almendra (1967-1969)", " Los Larkings", " Los Masters",
" Los Mods", " Almendra (1967-1969)", " Los Larkings", " Los Larkings",
" Los Larkings", " Los Masters", " Los Mods", " Los Masters",
" Los Mods", " Los Sbirros", " Los Sbirros", " Los Sbirros",
" Los Sbirros", " Almendra (1967-1969)", " Los Sbirros")), class = "data.frame", row.names = c(NA,
-25L))
data %>%
select(from = persona, to = grupo, grupo=grupo, name=persona) -> prepared.data
kable(head(prepared.data))
| from | to | grupo | name |
|---|---|---|---|
| Luis Alberto Spinetta | Bundleman | Bundleman | Luis Alberto Spinetta |
| Luis Alberto Spinetta | Los Larkings | Los Larkings | Luis Alberto Spinetta |
| Luis Alberto Spinetta | Los Masters | Los Masters | Luis Alberto Spinetta |
| Luis Alberto Spinetta | Los Mods | Los Mods | Luis Alberto Spinetta |
| Luis Alberto Spinetta | Los Sbirros | Los Sbirros | Luis Alberto Spinetta |
| Luis Alberto Spinetta | Almendra (1967-1969) | Almendra (1967-1969) | Luis Alberto Spinetta |
La gráfica
tbl_graph(edges=prepared.data, directed = TRUE) %>%
ggraph(layout = "linear") +
geom_edge_arc(aes(color=grupo), edge_width=1.5, edge_alpha = 0.5, fold = TRUE,) +
geom_node_point(size = 2, color="#67a9cf") +
geom_node_text(aes(label = str_wrap(name,13)), size = 3, nudge_y =-.7, angle = 90, fontface = "bold", hjust=.5) +
coord_cartesian(clip = "off") +
theme_elegante_std(base_family = "Assistant") +
theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
legend.position = "none") +
labs(title = "Historia musical de Luis Albero Spinetta",
subtitle = "Los comienzos"
)
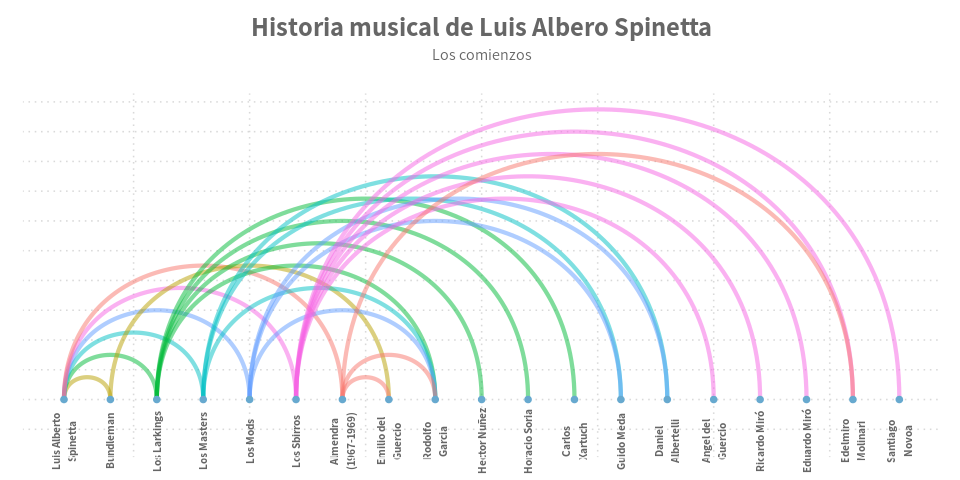
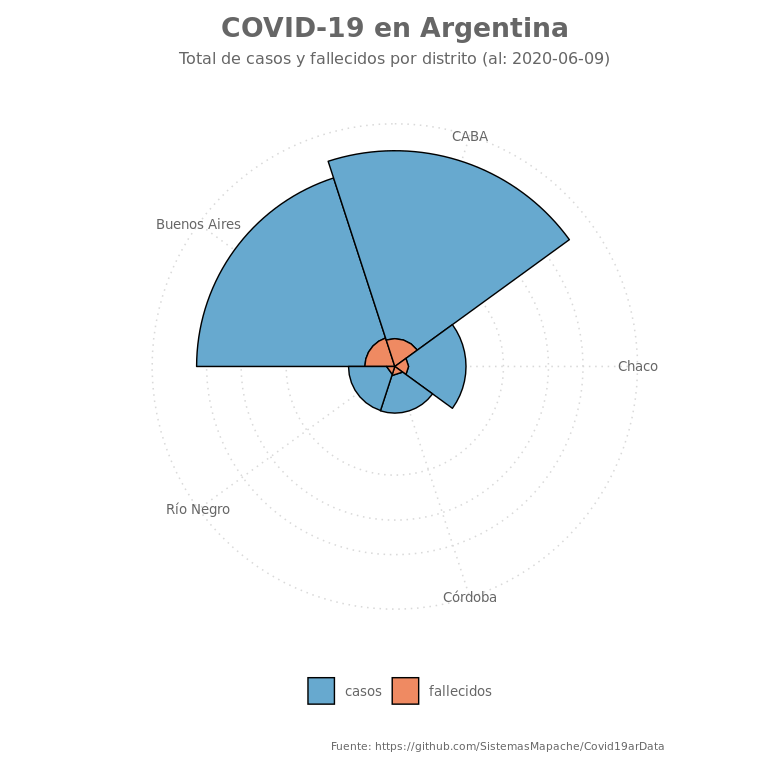
Día 6: Un gráfico de aros de cebolla
Odio las donas, pero bueno, en realidad se llaman “donuts plots”, una variante de un clásico gráfico de torta, solo sin el centro, a diferencia de los últimos, los gráficos de donas, al no mostrar el área completa, logran que el usuario se enfoque más en la proporción de cada sector, lo cual mejora la percepción de los cambios. Además, eventualmente permiten usar el área del centro para agregar más información.
Los datos
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
break_porc <- .95
covid.data %>%
select(distrito=osm_admin_level_4, casos=nue_casosconf_diff) %>%
group_by(distrito) %>%
summarise(casos=sum(casos)) %>%
mutate(porc = casos / sum(casos)) %>%
ungroup() %>%
arrange(-porc) %>%
mutate(cporc = cumsum(porc),
distrito = ifelse(cporc < break_porc, distrito, 'Resto')) %>%
group_by(distrito) %>%
summarise(casos = sum(casos),
porc = sum(round(porc*100,2))) -> data
data$porc[data$distrito == 'Resto'] <- 100 - sum(data$porc[data$distrito != 'Resto'])
data %>%
mutate( ymax = cumsum(porc),
ymin = lag(ymax, default=0)
) -> data
mac_perc <- sum(data$porc[data$distrito != 'Resto'])
data$distrito <- with(data, reorder(distrito, porc))
kable(head(data))
| distrito | casos | porc | ymax | ymin |
|---|---|---|---|---|
| Buenos Aires | 9590 | 38.73 | 38.73 | 0.00 |
| CABA | 11965 | 48.32 | 87.05 | 38.73 |
| Chaco | 1118 | 4.51 | 91.56 | 87.05 |
| Resto | 1598 | 6.46 | 98.02 | 91.56 |
| Río Negro | 491 | 1.98 | 100.00 | 98.02 |
La gráfica
data %>%
ggplot(aes(ymax=ymax, ymin=ymin, xmax=4, xmin=3, fill=distrito)) +
geom_rect(color="gray90", size=1.2) +
geom_label_repel(mapping = aes(x=3.5, y=ymin + (ymax - ymin)/2,
colour = ifelse(porc > 10, 2, 0),
label = paste0(distrito, ": ", format(porc, digits=2, trim=FALSE), "%\nCasos:",
format(casos, big.mark = ",", trim=FALSE))),
family = "Assistant",
nudge_y = 1,
nudge_x = 1) +
coord_polar(theta="y") + # Try to remove that to understand how the chart is built initially
xlim(c(2, 4)) +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Distribución del ", mac_perc , "% de los casos por distrito\n (Datos al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData"
) +
theme_elegante_std(base_family = "Assistant") +
theme(axis.title.x=element_blank(),
axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
legend.position = "none") +
scale_fill_brewer(palette = "Blues")
Día 7: Gráficos “Ridgeline”
Los graficos “ridgeline” mapean la distribución de múltiples variables continuas con un conjunto de variables categoricas en la forma de curvas “suaves” que permite comparar cada categoría:
Los datos:
library("tidyverse")
library("ggridges")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
break_porc <- .95
covid.data %>%
mutate(distrito = osm_admin_level_4,
casos = nue_casosconf_diff) %>%
select(distrito, casos) -> data
data %>%
group_by(distrito) %>%
summarise(casos=sum(casos)) %>%
mutate(porc = casos / sum(casos)) %>%
arrange(-porc) %>%
mutate(cporc = cumsum(porc),
distrito = ifelse(cporc < break_porc, distrito, 'Resto')) %>%
group_by(distrito) %>%
summarise(casos = sum(casos),
porc = sum(round(porc*100,2))) -> principales
perc <- sum(principales$porc[principales$distrito != 'Resto'])
data %>%
left_join(principales, by="distrito") %>%
mutate(distrito = ifelse(is.na(casos.y), 'Resto', distrito)) %>%
select(distrito, casos=casos.x) -> plot_data
kable(head(plot_data))
| distrito | casos |
|---|---|
| CABA | 1 |
| Resto | 0 |
| Resto | 0 |
| Buenos Aires | 1 |
| Resto | 0 |
| Resto | 0 |
La gráfica:
plot_data %>%
ggplot(aes(x = casos, y = distrito, fill = distrito)) +
geom_density_ridges() +
theme(legend.position = "none") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Distribución de casos diarios (al: ", last_date, ")\nDetalle en los distritos que suman el ", perc, "% de los casos totales del país") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Distritos",
x = "Cantidades de casos diarios"
) +
scale_x_continuous(breaks = c(c(0,5, 10, 20, 50), seq(from=75, to=max(data$casos)+25, by = 25))) +
theme_elegante_std(base_family = "Assistant") +
theme(legend.position = "none")
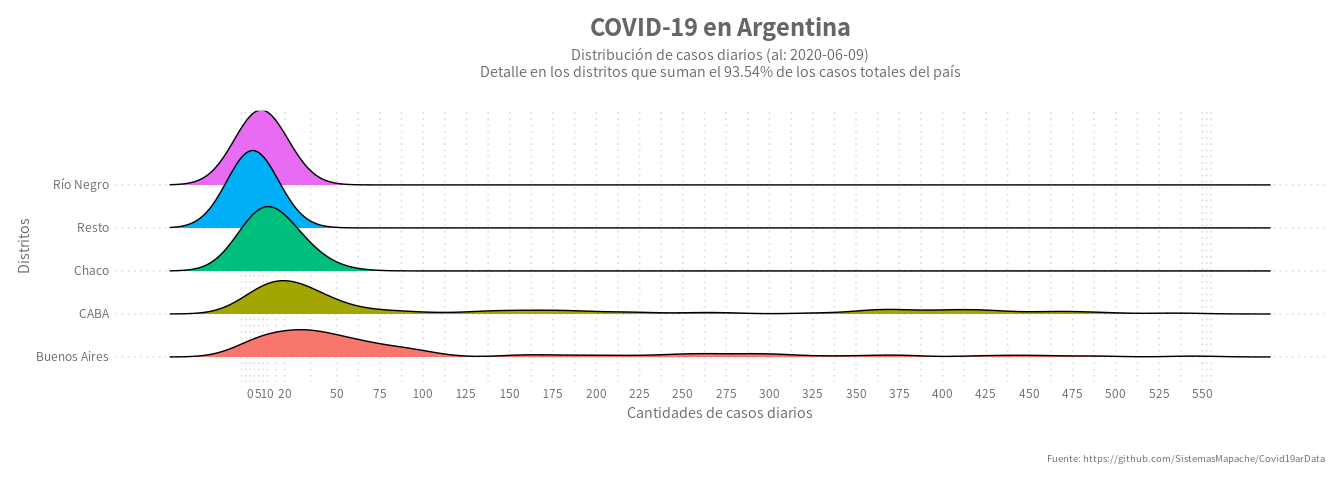
Día 8: Gráfico de contornos
Este tipo de gráficos básicamente une puntos x e y que comparte un mismo valor de una tercer variable z, el caso típico, son los mapa cartográficos , cuyas lineas unen puntos de semejante altura.
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Para descarga de los datos actualizados
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# hdi <- read.csv("https://data.humdata.org/dataset/05b5d8f1-9e7f-4379-9958-125c203d12ac/resource/4a7fd374-7e35-4c04-b7c8-25e5943aa476/downlo
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
hdi <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/hdi.Rda","rb"))
hdi %>%
group_by(country_code) %>%
arrange(year) %>%
slice(n()) %>%
select(country_code, country, year, value) -> last_hdi
last_date <- max(as.Date(covid.data$dateRep,"%d/%m/%Y"))
paises_de_interes <- c( 'Argentina',
"Niger", "Norway", "EEUU", "Mauritania")
covid.data %>%
group_by(countriesAndTerritories, countryterritoryCode) %>%
summarize(casos = sum(cases), fallecidos = sum(deaths)) %>%
ungroup() %>%
inner_join(last_hdi,
by = c("countryterritoryCode" = "country_code")
) %>%
mutate(pais = ifelse(countriesAndTerritories == 'United_States_of_America', 'EEUU', countriesAndTerritories)) %>%
select(pais, casos, fallecidos, HDI = value) %>%
mutate(pais_etiquetado = ifelse(pais %in% paises_de_interes, paste0(pais, " (casos: ", format(casos, digits=0, big.mark = ',', trim=TRUE), " hdi: ", HDI, ")"), NA)) -> plot_data
kable(head(plot_data))
| pais | casos | fallecidos | HDI | pais_etiquetado |
|---|---|---|---|---|
| Afghanistan | 27532 | 546 | 0.468 | NA |
| Albania | 1788 | 39 | 0.716 | NA |
| Algeria | 11385 | 811 | 0.717 | NA |
| Andorra | 855 | 52 | 0.830 | NA |
| Angola | 155 | 7 | 0.526 | NA |
| Antigua_and_Barbuda | 26 | 3 | 0.774 | NA |
La gráfica:
plot_data %>%
ggplot(aes(x=HDI, y=casos)) +
geom_point(color = "#67a9cf", alpha=.5, size=3) +
geom_smooth(method = 'lm',formula='y ~ x', se=FALSE, color="#ef8a62") +
geom_density2d(contour = TRUE, n = 1000) +
geom_label_repel(mapping = aes(label = pais_etiquetado),
color="#67a9cf",family = "Assistant", vjust = -1.2, hjust = 1.1, fontface="bold") +
scale_y_log10(
breaks = scales::trans_breaks("log10", function(x) 10^x),
labels = scales::trans_format("log10", scales::math_format(10^.x))
) +
labs(title = paste("COVID-19"),
subtitle = paste0("¿Hay relación entre el desarrollo humano y la cantidad de infecciones?\n (Datos al: ", last_date, ")") ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "log10(Cantidad de infectados)",
x = "Human development Index (2013)"
) +
theme_elegante_std(base_family = "Assistant")
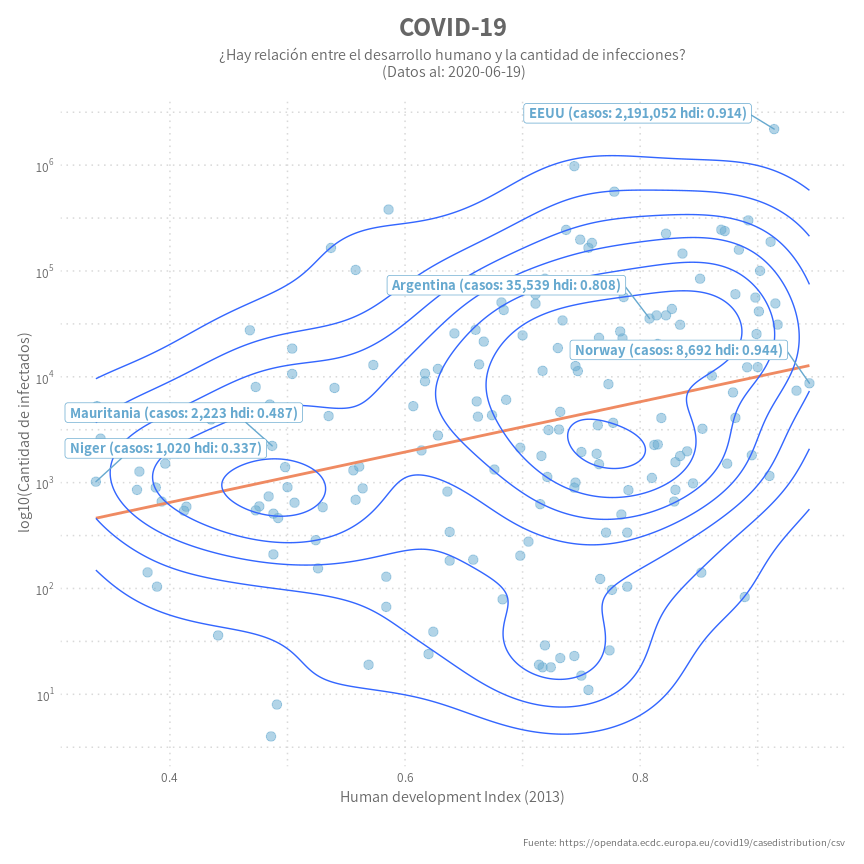
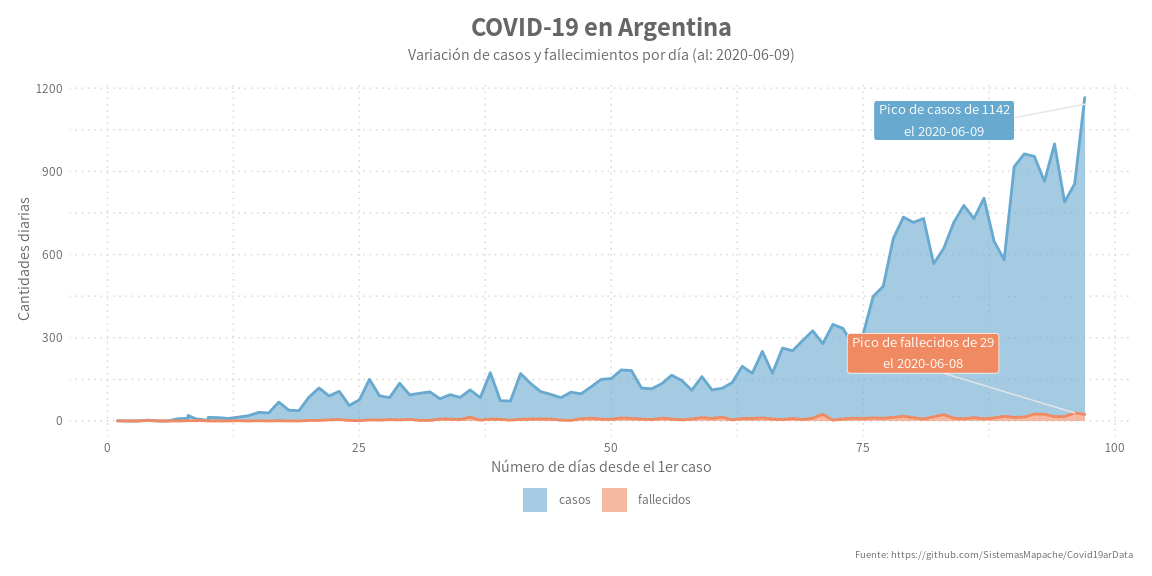
Día 9: Areas apiladas
Es una variante del gráfico de areas, que a su vez es una variante del de lineas, solo que en este caso se “apilan” más de una variable, lo que es útil para compararlas.
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
covid.data %>%
mutate(fecha=as.Date(fecha,"%d/%m/%Y")) %>%
group_by(dia_inicio,fecha) %>%
summarize(casos = sum(nue_casosconf_diff),
fallecidos = sum(nue_fallecidos_diff)) %>%
select(dia = dia_inicio, fecha, casos, fallecidos) %>%
pivot_longer(-c("dia", "fecha"), names_to = 'metrica', values_to='cantidades') -> data
data %>%
left_join(data %>%
group_by(metrica) %>%
summarise(cantidades = max(cantidades), maximo = TRUE),
by = c("metrica", "cantidades")
) %>%
mutate(maximo = ifelse(maximo, paste0("Pico de ", metrica, " de ", cantidades, "\nel ", fecha), NA)) -> plot_data
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
kable(head(plot_data))
| dia | fecha | metrica | cantidades | maximo |
|---|---|---|---|---|
| 1 | 2020-03-02 | casos | 1 | NA |
| 1 | 2020-03-02 | fallecidos | 0 | NA |
| 2 | 2020-03-03 | casos | 0 | NA |
| 2 | 2020-03-03 | fallecidos | 0 | NA |
| 3 | 2020-03-04 | casos | 0 | NA |
| 3 | 2020-03-04 | fallecidos | 0 | NA |
La gráfica:
plot_data %>%
ggplot(aes(x=dia, y=cantidades, fill=metrica, color=metrica)) +
geom_area(alpha=0.6 , size=1) +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Variación de casos y fallecimientos por día (al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Cantidades diarias",
x = "Número de días desde el 1er caso"
) +
geom_label_repel(mapping = aes(label = maximo),
color = "white",
segment.color="gray90",
family = "Assistant",
vjust = -1,
hjust = 1.5,
box.padding = 1,
show.legend = FALSE) +
scale_fill_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
scale_color_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
guides(color = FALSE, label=FALSE) +
theme_elegante_std(base_family = "Assistant")
Día 10: Exploración de paletas de colores
Nada del otro mundo, un script para generar una gráfica de ejemplo de una paleta determinada de colores, en este ejemplo explorando la del paquete [wesanderson]
library("tidyverse")
library("wesanderson")
library("gridExtra")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
colores <- 20
paletas <- names(wes_palettes)
data.frame(x=factor(1:colores), y=1) %>%
ggplot(aes(x = x, y = y, fill = x)) +
geom_col() +
theme_elegante_std(base_family = "Assistant") +
theme(axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
legend.position = "none") -> g
plots <- list()
for (p in paletas) {
plots[[p]] <- g + scale_fill_manual(values = wes_palette(colores, name = p, type = "continuous"), name = "") +
labs(x = p)
}
grid.arrange(grobs = plots, ncol = 2)

Día 11: Mapa de Calor
Un mapa de calor muestra la variación en una determinada variable (por lo general continua) mediante el uso del color. En este ejemplo mapeamos tres variables, el distrito o provincia (categórica), la cantidad de días desde el primer contagio (categórica) y la variable continua de la cantidad de casos que es la que mapeamos a la dimensión del color.
library("tidyverse")
library("zoo")
library("forcats")
library("scales")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
first_date <- min(as.Date(covid.data$fecha,"%d/%m/%Y"))
ndias <- max(covid.data$dia_inicio)
dias <- expand.grid(distrito = unique(covid.data$osm_admin_level_4),
dia = 1:ndias,
stringsAsFactors = FALSE)
dias$fecha = first_date + dias$dia - 1
dias$casos = 0
dias_rolling <- 3
dias_ventana <- 14
dias %>%
left_join(covid.data, by=c("distrito" = "osm_admin_level_4", "dia" = "dia_inicio")) %>%
mutate(casos = replace_na(nue_casosconf_diff, 0)) %>%
select(dia, distrito, fecha=fecha.x, casos) %>%
group_by(dia, distrito, fecha) %>%
summarise(casos = sum(casos)) %>%
arrange(distrito, dia) %>%
group_by(distrito) %>%
filter(dia >= ndias - dias_ventana - dias_rolling,
distrito != 'Indeterminado') %>%
mutate(roll_casos_3 = rollmean(casos, dias_rolling, align='right', fill=0),
s_roll_casos_3 = replace_na((roll_casos_3-min(roll_casos_3))/(max(roll_casos_3)-min(roll_casos_3)),0),
label_casos = casos
) %>%
filter(dia >= ndias - dias_ventana ) -> plot_data
kable(head(plot_data))
| dia | distrito | fecha | casos | roll_casos_3 | s_roll_casos_3 | label_casos |
|---|---|---|---|---|---|---|
| 83 | Buenos Aires | 2020-05-23 | 236 | 197.0000 | 0.4371302 | 236 |
| 84 | Buenos Aires | 2020-05-24 | 315 | 236.6667 | 0.5251479 | 315 |
| 85 | Buenos Aires | 2020-05-25 | 299 | 283.3333 | 0.6286982 | 299 |
| 86 | Buenos Aires | 2020-05-26 | 273 | 295.6667 | 0.6560651 | 273 |
| 87 | Buenos Aires | 2020-05-27 | 296 | 289.3333 | 0.6420118 | 296 |
| 88 | Buenos Aires | 2020-05-28 | 254 | 274.3333 | 0.6087278 | 254 |
La gráfica:
plot_data %>%
ggplot(aes(x = dia, y = fct_reorder(distrito, casos), fill = s_roll_casos_3)) +
geom_tile(colour="gray80", size=0.2) +
geom_text(aes(label=label_casos, color = s_roll_casos_3 > .7)) +
scale_color_manual(guide = FALSE, values = c("gray30", "gray90")) +
scale_fill_distiller(palette = "YlGnBu", direction = 1, na.value = "white") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Evolución diaria de los casos por distrito (últimos ", dias_ventana, " días al: ", last_date, ")\n",
"Los números son casos diarios, la escala de color en función del promedio de casos de los últimos ", dias_rolling, " días") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
x = "Últimos días"
) +
theme_elegante_std(base_family = "Assistant") +
scale_x_continuous(breaks = seq(ndias - dias_ventana, ndias, by = 1)) +
theme(axis.title.y=element_blank(),
legend.position = "none")
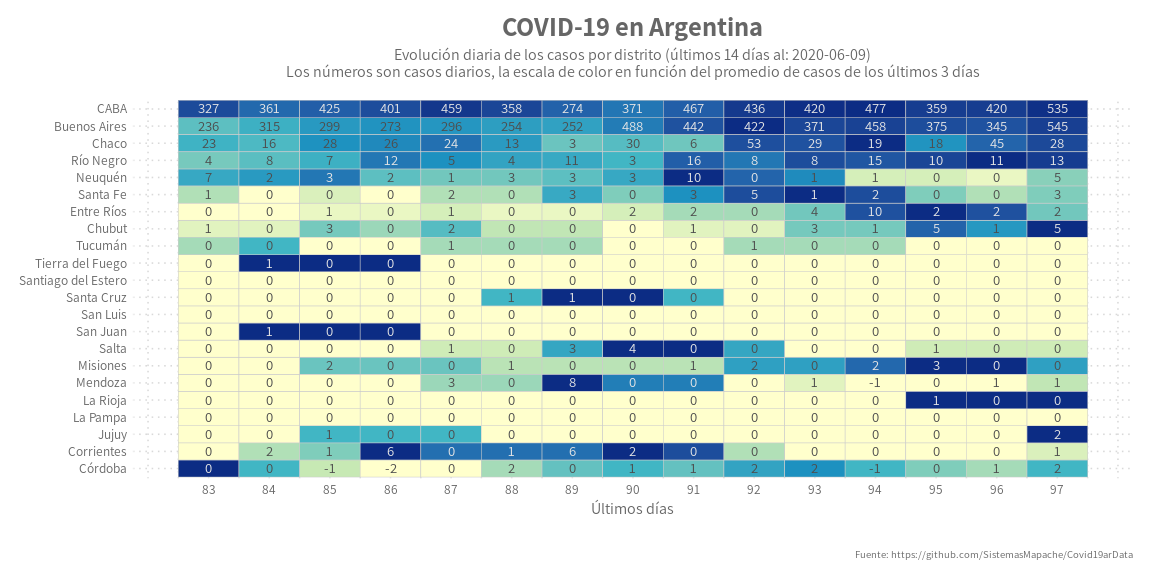
Día 12: Un gráfico lolipop
Es básicamente un gráfico de barras, pero se remplaza la barra por una línea que se completa con un punto al final dela barra.
library("tidyverse")
library("forcats")
library("countrycode")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Para descarga de los datos actualizados
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# hdi <- read.csv("https://data.humdata.org/dataset/05b5d8f1-9e7f-4379-9958-125c203d12ac/resource/4a7fd374-7e35-4c04-b7c8-25e5943aa476/downlo
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
last_date <- max(as.Date(covid.data$dateRep,"%d/%m/%Y"))
covid.data %>%
group_by(countryterritoryCode) %>%
summarize(casos = sum(cases), fallecidos = sum(deaths)) %>%
arrange(-casos) %>%
mutate(nr = row_number()) %>%
select(code_pais = countryterritoryCode, casos, fallecidos, nr) -> data
media_casos <- mean(data$casos)
media_fallecidos <- mean(data$fallecidos)
data %>%
filter(nr <= 50) %>%
mutate(sobre_media = casos > media_casos,
pais = paste0(countrycode(code_pais, origin = 'iso3c', destination = 'un.name.es'), " (", nr, ")")) -> plot_data
kable(head(plot_data))
| code_pais | casos | fallecidos | nr | sobre_media | pais |
|---|---|---|---|---|---|
| USA | 2191052 | 118434 | 1 | TRUE | Estados Unidos de América (1) |
| BRA | 978142 | 47748 | 2 | TRUE | Brasil (2) |
| RUS | 561091 | 7790 | 3 | TRUE | Federación de Rusia (3) |
| IND | 380532 | 12573 | 4 | TRUE | India (4) |
| GBR | 300469 | 42288 | 5 | TRUE | Reino Unido de Gran Bretaña e Irlanda del Norte (5) |
| ESP | 245268 | NA | 6 | TRUE | España (6) |
La gráfica:
plot_data %>%
ggplot(aes(x=fct_reorder(pais, -nr), y=casos, color = sobre_media)) +
geom_segment(aes( y=0 , xend = pais, yend = casos)) +
geom_point() +
coord_flip() +
geom_text(aes(label = format(abs(casos), digits=0, big.mark = ','),
vjust = .5,
hjust = -.1),
position = position_dodge(width=1)) +
labs(title = paste("COVID-19 en el mundo"),
subtitle = paste("Casos por pais al: ", last_date, '\nLos primeros 50 países') ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "Casos",
x = ""
) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5), limits = c(0, max(data$casos) * 1.01),
labels = scales::comma) +
scale_color_manual(labels = c("Sobre la media", "Debajo de la media"), values = c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Assistant")
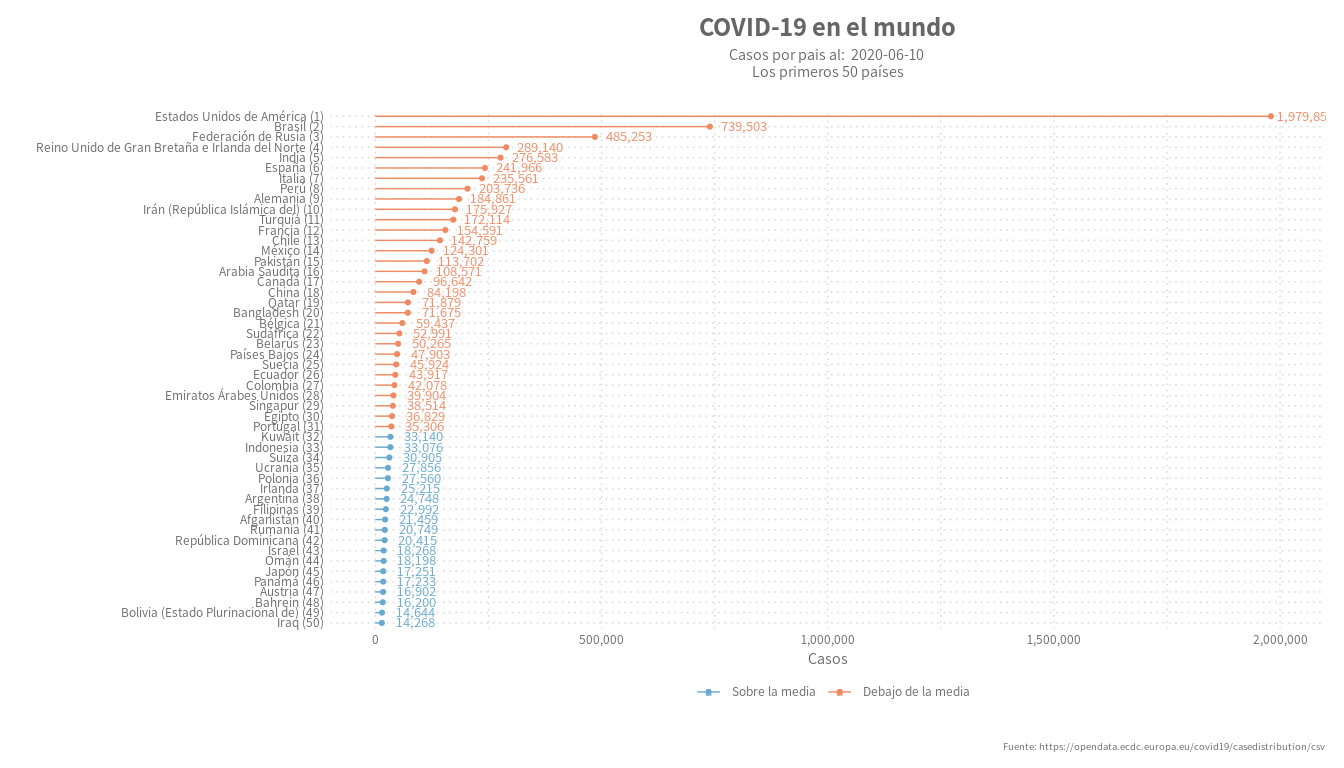
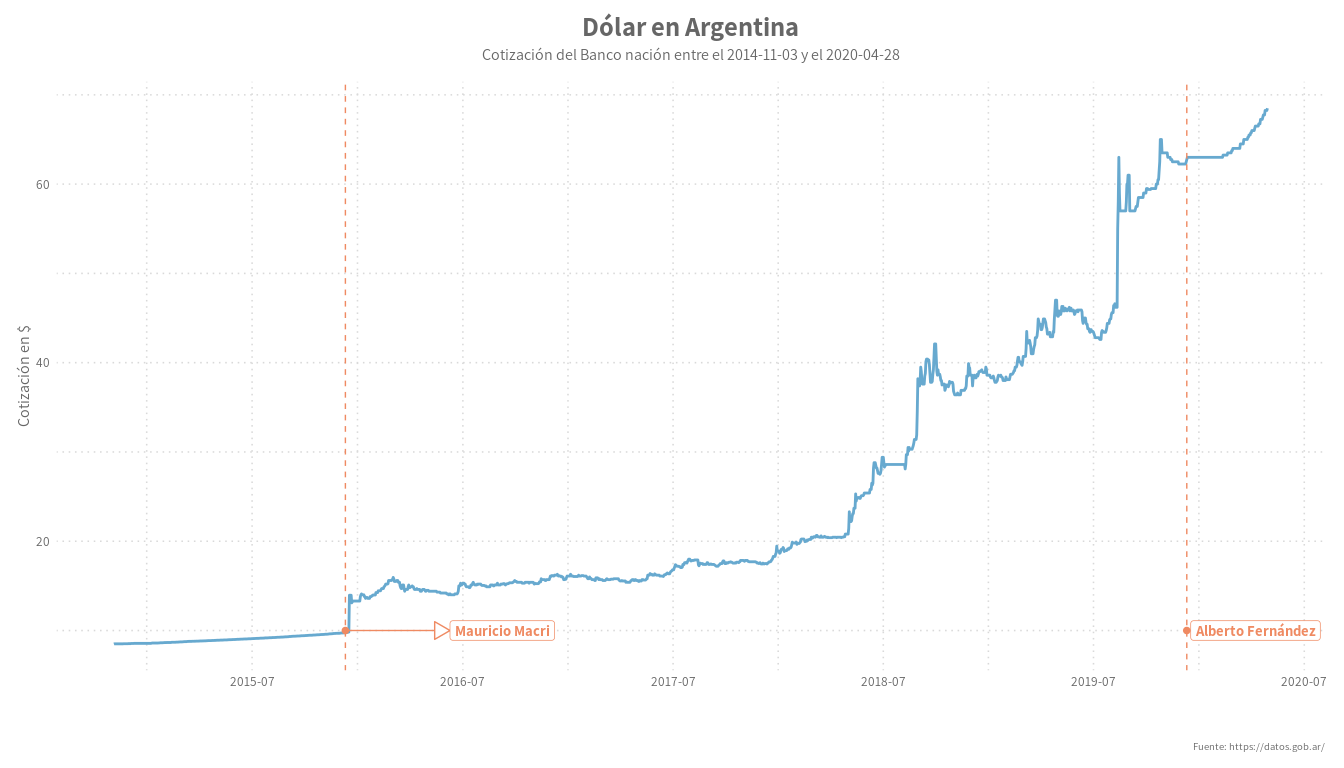
Día 13: Serie temporal
Nada del otro mundo, el clásico gráfico de lineas, en el eje X tenemos el tiempo y en el Y un precio, en este caso el vaalor del dólar en Argentina en los últimos años, con algunas anotaciones, como ser los comiencos de cada presidencia.
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
presidencias <- data.frame(fecha = as.Date(c('2019-12-10', '2015-12-10')), presidencia=c('Alberto Fernández', 'Mauricio Macri'))
# dolar <- read.csv("https://apis.datos.gob.ar/series/api/series/?ids=168.1_T_CAMBIOR_D_0_0_26&limit=5000&format=csv", na.strings = "", fileEncoding = "UTF-8-BOM",
# stringsAsFactors = FALSE)
dolar <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/dolar.Rda","rb"))
dolar$indice_tiempo = as.Date(dolar$indice_tiempo, format = "%Y-%m-%d")
kable(head(dolar))
| indice_tiempo | tipo_cambio_bna_vendedor |
|---|---|
| 2014-11-03 | 8.49 |
| 2014-11-04 | 8.51 |
| 2014-11-05 | 8.51 |
| 2014-11-06 | 8.51 |
| 2014-11-07 | 8.51 |
| 2014-11-08 | 8.51 |
La gráfica:
dolar %>%
ggplot(mapping = aes(x=indice_tiempo, y=tipo_cambio_bna_vendedor)) +
geom_line(size = 1, color="#67a9cf") +
geom_vline(data = presidencias,
mapping = aes(xintercept=fecha),
color = "#ef8a62",
linetype="dashed") +
geom_point(data = presidencias,
mapping = aes(x=fecha, y=10),
color = "#ef8a62",
size = 2) +
geom_label_repel(data = presidencias,
mapping = aes(x=fecha, y=10, label = presidencia),
hjust= -1,
color = "#ef8a62",
family = "Assistant", fontface = 'bold',
arrow = arrow(length = unit(0.03, "npc"), type = "closed", ends = "first")
) +
labs(title = paste("Dólar en Argentina"),
subtitle = paste("Cotización del Banco nación entre el", min(dolar$indice_tiempo), "y el" , max(dolar$indice_tiempo)),
caption = "Fuente: https://datos.gob.ar/",
y = "Cotización en $",
x = ""
) +
scale_x_date(date_breaks = "12 month", date_labels="%Y-%m") +
theme_elegante_std(base_family = "Assistant")
Día 12: Treemap
Los diagramas de árbol o “treemap” mapean dos variables, una categorica y otra cuantitativa, visualmente se construyen áreas por cada categoría con una superficie consistente con la variable numérica. En este ejemplo mapeamos la cantidad de casos acumulada de COVID por provincia, destacando las provincias que suman 95% de los casos totales
library("tidyverse")
library("ggrepel")
library("treemapify")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
first_date <- min(as.Date(covid.data$fecha,"%d/%m/%Y"))
ndias <- max(covid.data$dia_inicio)
dias <- expand.grid(distrito = unique(covid.data$osm_admin_level_4),
dia = 1:ndias,
stringsAsFactors = FALSE)
dias %>%
left_join(covid.data, by=c("distrito" = "osm_admin_level_4", "dia" = "dia_inicio")) %>%
mutate(casos = replace_na(nue_casosconf_diff, 0)) %>%
select(distrito, casos) %>%
filter(distrito != 'Indeterminado') %>%
group_by(distrito) %>%
summarise(casos = sum(casos)) %>%
arrange(-casos) %>%
mutate(porc = cumsum(casos/sum(casos))) -> plot_data
kable(head(plot_data))
| distrito | casos | porc |
|---|---|---|
| CABA | 11965 | 0.4832001 |
| Buenos Aires | 9590 | 0.8704870 |
| Chaco | 1118 | 0.9156369 |
| Río Negro | 491 | 0.9354656 |
| Córdoba | 466 | 0.9542848 |
| Santa Fe | 279 | 0.9655521 |
La gráfica:
plot_data %>%
ggplot(aes(area = casos, fill = casos, label=paste0(distrito, ": ", format(casos, big.mark = ".", decimal.mark = ",")),
subgroup = ifelse(porc <= .95, "95%", ""))) +
geom_treemap() +
geom_treemap_subgroup_border() +
geom_treemap_subgroup_text(place = "centre", grow = T, alpha = .5, colour =
"White", fontface = "italic", min.size = 0) +
geom_treemap_text(place = "middle", grow = T, reflow = T, alpha = 0.8, colour = "black", family= "Assistant",
padding.x = grid::unit(3, "mm"),
padding.y = grid::unit(3, "mm")) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Distribución de los casos por distritos al: ", last_date),
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData"
) +
scale_fill_distiller(palette = "YlGnBu", direction = 1, na.value = "white") +
theme(legend.position = "none")

Día 15: Dendograma
library("tidyverse")
library("ggdendro")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
dendro_data_k <- function(hc, k) {
hcdata <- ggdendro::dendro_data(hc, type = "rectangle")
seg <- hcdata$segments
labclust <- cutree(hc, k)[hc$order]
segclust <- rep(0L, nrow(seg))
heights <- sort(hc$height, decreasing = TRUE)
height <- mean(c(heights[k], heights[k - 1L]), na.rm = TRUE)
for (i in 1:k) {
xi <- hcdata$labels$x[labclust == i]
idx1 <- seg$x >= min(xi) & seg$x <= max(xi)
idx2 <- seg$xend >= min(xi) & seg$xend <= max(xi)
idx3 <- seg$yend < height
idx <- idx1 & idx2 & idx3
segclust[idx] <- i
}
idx <- which(segclust == 0L)
segclust[idx] <- segclust[idx + 1L]
hcdata$segments$clust <- segclust
hcdata$segments$line <- as.integer(segclust < 1L)
hcdata$labels$clust <- labclust
hcdata
}
set_labels_params <- function(nbLabels,
direction = c("tb", "bt", "lr", "rl"),
fan = FALSE) {
if (fan) {
angle <- 360 / nbLabels * 1:nbLabels + 90
idx <- angle >= 90 & angle <= 270
angle[idx] <- angle[idx] + 180
hjust <- rep(0, nbLabels)
hjust[idx] <- 1
} else {
angle <- rep(0, nbLabels)
hjust <- 0
if (direction %in% c("tb", "bt")) { angle <- angle + 45 }
if (direction %in% c("tb", "rl")) { hjust <- 1 }
}
list(angle = angle, hjust = hjust, vjust = 0.5)
}
plot_ggdendro <- function(hcdata,
direction = c("lr", "rl", "tb", "bt"),
fan = FALSE,
scale.color = NULL,
branch.size = 1,
label.size = 3,
nudge.label = 0.01,
expand.y = 0.1,
nbreaks = 10) {
direction <- match.arg(direction) # if fan = FALSE
ybreaks <- pretty(segment(hcdata)$y, n = nbreaks)
ymax <- max(segment(hcdata)$y)
## branches
p <- ggplot() +
geom_segment(data = segment(hcdata),
aes(x = x,
y = y,
xend = xend,
yend = yend,
linetype = factor(line),
colour = factor(clust)),
lineend = "round",
show.legend = FALSE,
size = branch.size)
## orientation
if (fan) {
p <- p +
coord_polar(direction = -1) +
scale_x_continuous(breaks = NULL,
limits = c(0, nrow(label(hcdata)))) +
scale_y_reverse(breaks = ybreaks, labels = scales::comma)
} else {
p <- p + scale_x_continuous(breaks = NULL)
if (direction %in% c("rl", "lr")) {
p <- p + coord_flip()
}
if (direction %in% c("bt", "lr")) {
p <- p + scale_y_reverse(breaks = ybreaks, labels = scales::comma)
} else {
p <- p + scale_y_continuous(breaks = ybreaks, labels = scales::comma)
nudge.label <- -(nudge.label)
}
}
# labels
labelParams <- set_labels_params(nrow(hcdata$labels), direction, fan)
hcdata$labels$angle <- labelParams$angle
p <- p +
geom_text(data = label(hcdata),
aes(x = x,
y = y,
label = label,
colour = factor(clust),
angle = angle),
vjust = labelParams$vjust,
hjust = labelParams$hjust,
nudge_y = ymax * nudge.label,
size = label.size,
show.legend = FALSE)
# colors and limits
if (!is.null(scale.color)) {
p <- p + scale_color_manual(values = scale.color)
}
ylim <- -round(ymax * expand.y, 1)
p <- p + expand_limits(y = ylim)
p
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
first_date <- min(as.Date(covid.data$fecha,"%d/%m/%Y"))
ndias <- max(covid.data$dia_inicio)
dias <- expand.grid(distrito = unique(covid.data$osm_admin_level_4),
dia = 1:ndias,
stringsAsFactors = FALSE)
dias %>%
left_join(covid.data, by=c("distrito" = "osm_admin_level_4", "dia" = "dia_inicio")) %>%
mutate(casos = replace_na(nue_casosconf_diff, 0)) %>%
select(distrito, casos) %>%
filter(distrito != 'Indeterminado') %>%
group_by(distrito) %>%
summarise(casos = sum(casos)) %>%
arrange(-casos) %>%
mutate(porc = cumsum(casos/sum(casos))) -> data
data_d <- as.data.frame(data[, 2])
rownames(data_d) <- data$distrito
D <- dist(data_d)
hc <- hclust(D)
hcdata <- dendro_data_k(hc, 5)
plot_ggdendro(hcdata,
direction = "rl",
label.size = 3,
branch.size = .8,
nudge.label = 0.02,
expand.y = 0.2,
nbreaks = 10) +
# scale_y_continuous(labels = scales::comma) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Agrupación de provincias por cantidad de casos en 5 grupos principales (al: ", last_date, ")"),
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Cantidad de casos",
x = "")
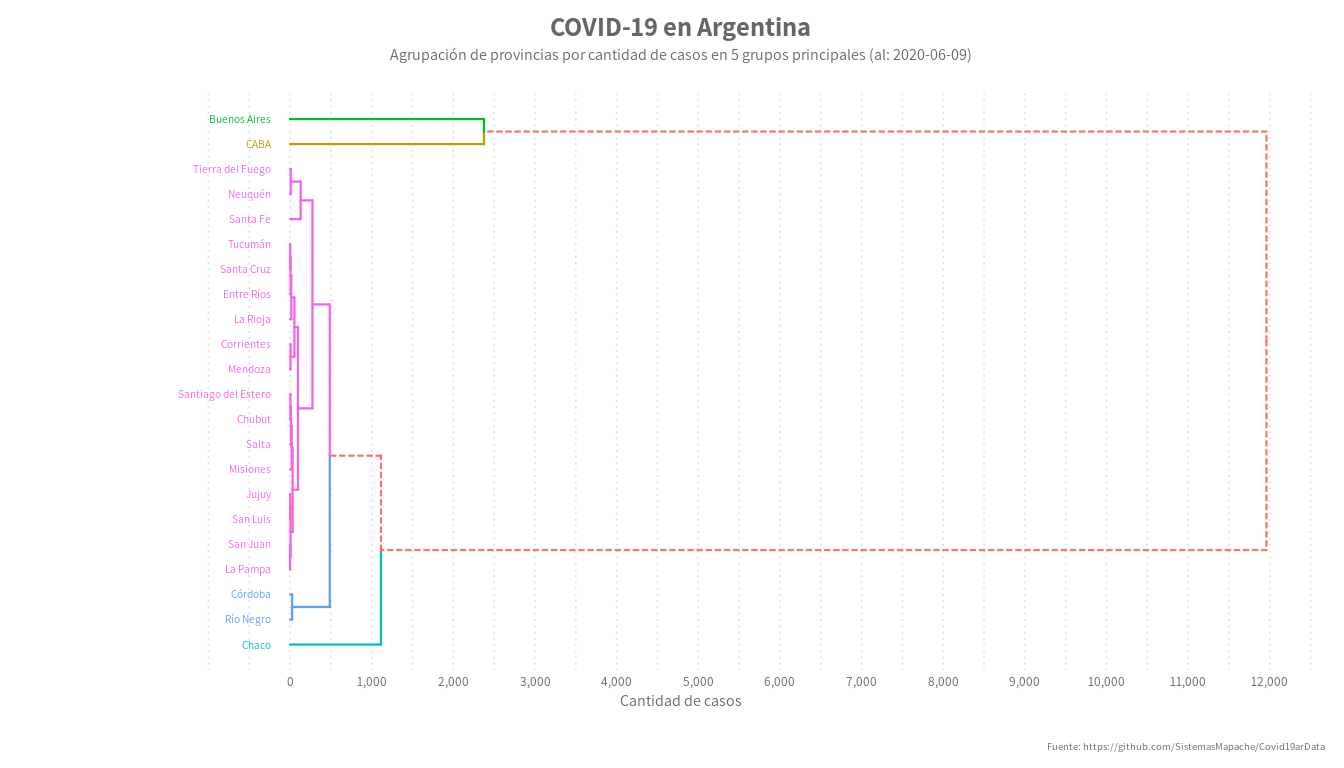
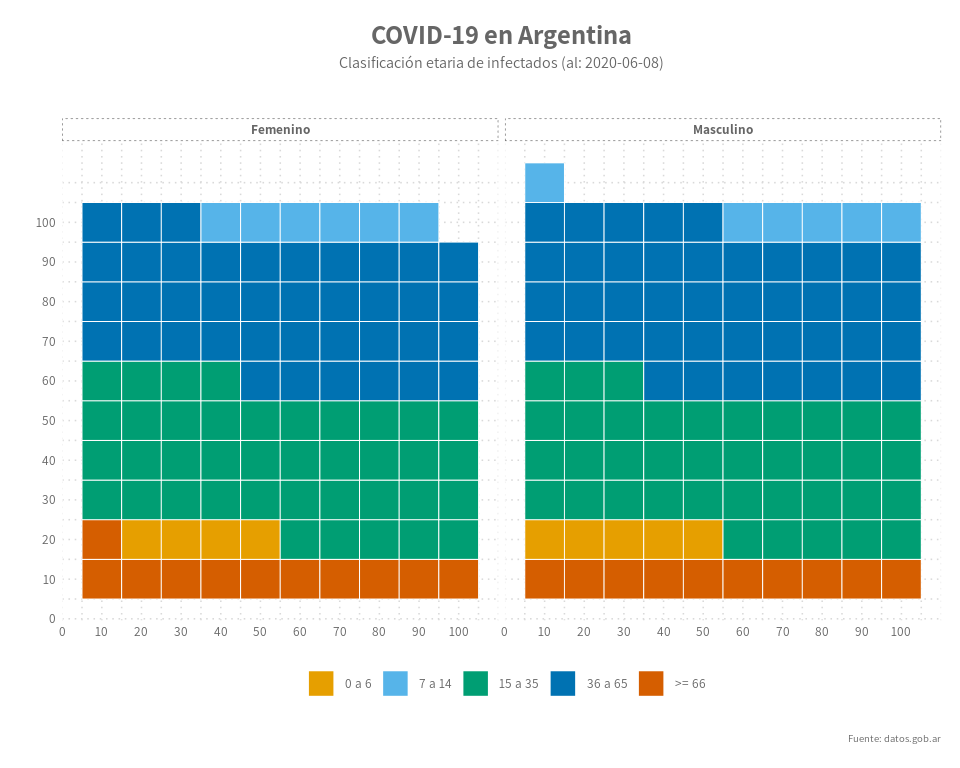
Día 16: Graficos de Waffle
Este gráfico, en su forma más básica, trabaja con una variable categoríca y una continua relacionada a la primera, normalmente una frecuencia. Es de la familia de los gráficos de torta, aunque, seguramente es un poco mejor para entender las diferencias entre los distintos niveles de una catgoría. Por lo general, se trabaja con una matriz de cuadrados de 10 x 10 que representa el 100% de la muestra a representar, cada categoría tendrá su porcentaje dentro de la muestra y el mismo se redondeará a múltiplos de 10 para asignar el numero de cuadrados que le va a tocar. Hay muchas variantes de este gráfico, dónde se usan otras formas, circulos, hexagonos o incluso pictogramas, pero la idea sigue siendo la misma.
En este ejemplo, vemos como se distribuyen los casos de COVID-19 en Argentina, facetado por sexo, en este caso cada cuadrado representa el 10% de su grupo y el 5% del total de casos.
library("tidyverse")
library("waffle")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read.csv(file='https://sisa.msal.gov.ar/datos/descargas/covid-19/files/Covid19Casos.csv', stringsAsFactors = FALSE, fileEncoding = "UTF-16")
# Datos reproducibles
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.arg.Rda","rb"))
last_date <- max(covid.data$fecha_apertura, na.rm = TRUE)
covid.data %>%
filter(clasificacion_resumen == 'Confirmado',
sexo != 'NR') %>%
mutate(internado = fecha_internacion != "",
cui = fecha_cui_intensivo != "",
arm = replace_na(asistencia_respiratoria_mecanica == "SI",FALSE),
fallecido = replace_na(fallecido == "SI", FALSE),
sexo = ifelse(sexo == 'M', 'Masculino', 'Femenino'),
internado = ifelse(internado, 'Internado', 'Ambulatorio'),
fallecido = ifelse(fallecido, 'Fallecido', 'Recuperado')) %>%
mutate(clasif_edad = case_when(edad <= 6 ~ '0 a 6',
edad > 6 & edad <= 14 ~ '7 a 14',
edad > 14 & edad <= 35 ~ '15 a 35',
edad > 35 & edad <= 65 ~ '36 a 65',
edad > 65 ~ '>= 66',
TRUE ~ 'Indeterminado')) %>%
select(sexo, edad, clasif_edad, internado, cui, arm, fallecido) %>%
group_by(sexo, clasif_edad) %>%
summarise(n = n()) %>%
mutate(freq = round(100*(n / sum(n)),0),
clasif_edad = factor(clasif_edad, c('0 a 6', '7 a 14', '15 a 35', '36 a 65', '>= 66'))) -> plot_data
kable(head(plot_data))
| sexo | clasif_edad | n | freq |
|---|---|---|---|
| Femenino | >= 66 | 1339 | 11 |
| Femenino | 0 a 6 | 519 | 4 |
| Femenino | 15 a 35 | 4504 | 39 |
| Femenino | 36 a 65 | 4583 | 39 |
| Femenino | 7 a 14 | 705 | 6 |
| Femenino | NA | 4 | 0 |
La grafica:
plot_data %>%
ggplot(aes(fill = clasif_edad, values = freq)) +
geom_waffle(n_rows = 10, size = 0.33, colour = "white", flip = TRUE) +
scale_y_continuous(breaks = seq(0, 10, by = 1), labels = function(x) x * 10) +
scale_x_continuous(breaks = seq(0, 10, by = 1), labels = function(x) x * 10) +
coord_equal() +
facet_wrap(~ sexo) +
scale_fill_manual(values=c("#E69F00", "#56B4E9", "#009E73", "#0072B2", "#D55E00", "#CC79A7")) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Clasificación etaria de infectados (al: ", last_date, ")\n"),
caption = "Fuente: datos.gob.ar",
y = "",
x = "")
# theme(axis.text.x=element_blank())
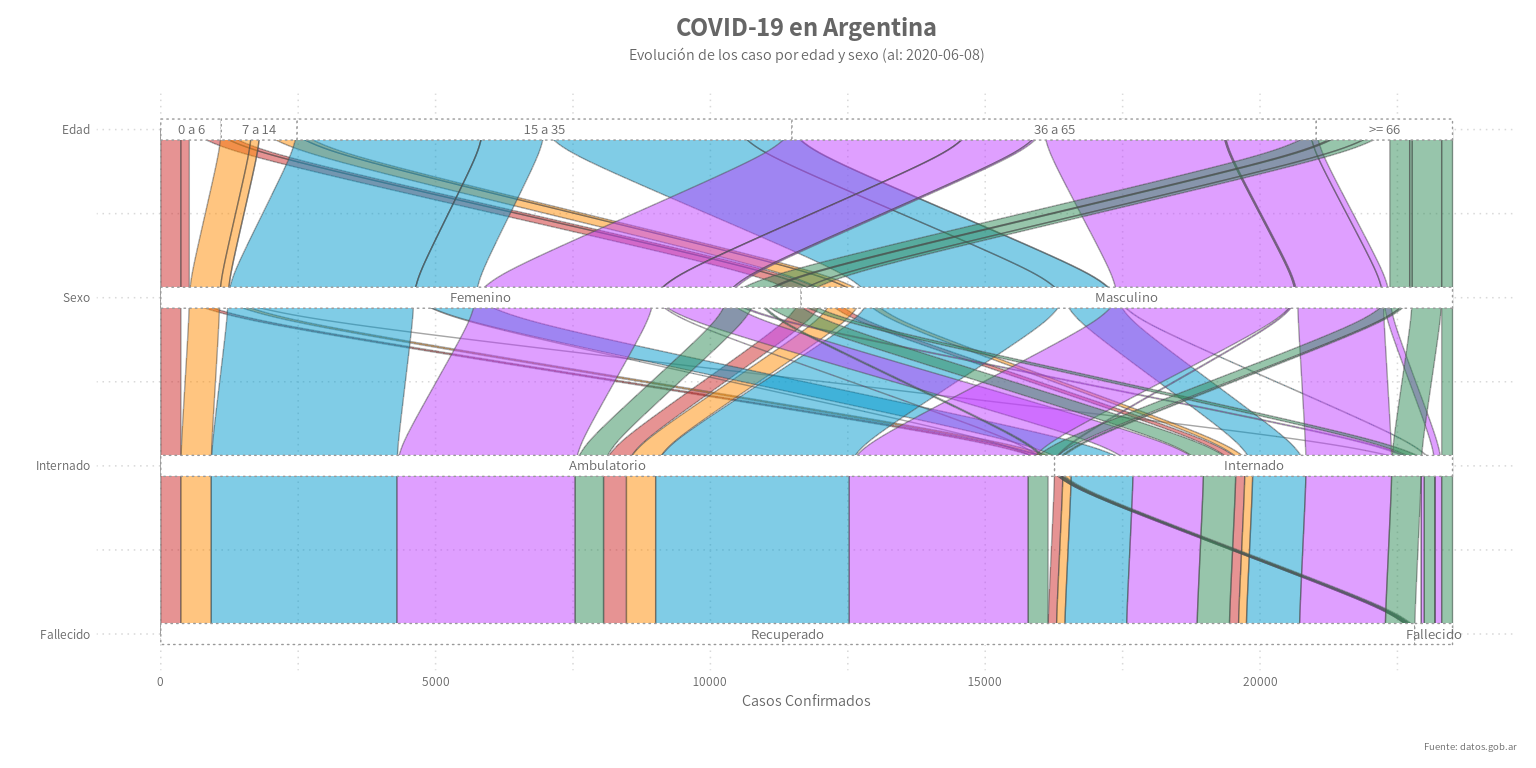
Día 17: Un Sankey o Alluvial
Los diagramas de Sankey muestran flujos entre distintas variables categorícas y sus cantidades en proporción entre sí. El ancho de las flechas o líneas se utiliza para mostrar sus magnitudes. Las flechas o líneas de flujo pueden combinarse o dividirse a través de sus trayectorias en cada etapa de un proceso. El color se suele utilizar para distingir mejor las diferentes categorías o para mostrar la transición de un estado del proceso a otro. Normalmente, los gráficos de Sankey se utilizan para mostrar visualmente la transferencia de energía, dinero o materiales, pero pueden usarse para mostrar el flujo de cualquier proceso aislado del sistema.
En este ejemplo, tomamos los casos de COVID-19 en Argentina y analizamos la evolución de los mismos. Los dos primeros niveles muestran como se componene los casos entre el sexo y el rango etario, y los siguientes muestran la evolución posterior de los casos.
library("tidyverse")
library("ggalluvial")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read.csv(file='https://sisa.msal.gov.ar/datos/descargas/covid-19/files/Covid19Casos.csv', stringsAsFactors = FALSE, fileEncoding = "UTF-16")
# Datos reproducibles
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.arg.Rda","rb"))
last_date <- max(covid.data$fecha_apertura, na.rm = TRUE)
covid.data %>%
filter(clasificacion_resumen == 'Confirmado',
!is.na(edad),
sexo != 'NR') %>%
mutate(internado = fecha_internacion != "",
cui = fecha_cui_intensivo != "",
arm = replace_na(asistencia_respiratoria_mecanica == "SI",FALSE),
fallecido = replace_na(fallecido == "SI", FALSE),
sexo = ifelse(sexo == 'M', 'Masculino', 'Femenino'),
internado = ifelse(internado, 'Internado', 'Ambulatorio'),
fallecido = ifelse(fallecido, 'Fallecido', 'Recuperado')) %>%
mutate(clasif_edad = case_when(edad <= 6 ~ '0 a 6',
edad > 6 & edad <= 14 ~ '7 a 14',
edad > 14 & edad <= 35 ~ '15 a 35',
edad > 35 & edad <= 65 ~ '36 a 65',
edad > 65 ~ '>= 66')) %>%
select(sexo, edad, clasif_edad, internado, cui, arm, fallecido) %>%
mutate(clasif_edad = factor(clasif_edad, c('0 a 6', '7 a 14', '15 a 35', '36 a 65', '>= 66')),
internado = factor(internado, c("Ambulatorio", "Internado")),
fallecido = factor(fallecido, c("Recuperado", "Fallecido"))
) %>%
group_by(clasif_edad, sexo, internado, fallecido) %>%
summarise(n = n()) %>%
ungroup() -> plot_data
kable(head(plot_data))
| clasif_edad | sexo | internado | fallecido | n |
|---|---|---|---|---|
| 0 a 6 | Femenino | Ambulatorio | Recuperado | 366 |
| 0 a 6 | Femenino | Internado | Recuperado | 153 |
| 0 a 6 | Masculino | Ambulatorio | Recuperado | 416 |
| 0 a 6 | Masculino | Internado | Recuperado | 165 |
| 7 a 14 | Femenino | Ambulatorio | Recuperado | 552 |
| 7 a 14 | Femenino | Internado | Recuperado | 152 |
La grafica:
plot_data %>%
ggplot(mapping=aes(y = n,
axis1 = fallecido, axis2 = internado, axis3 = sexo, axis4 = clasif_edad)) +
geom_alluvium(aes(fill = clasif_edad),
color = "Gray30",
width = 0, knot.pos = 0.1, reverse = FALSE) +
guides(fill = FALSE) +
geom_stratum(width = 1/8, reverse = FALSE, color="gray60", linetype = 3) +
geom_text(stat = "stratum", infer.label = TRUE, reverse = FALSE) +
scale_x_continuous(breaks = 1:4, labels = c("Fallecido", "Internado", "Sexo", "Edad")) +
coord_flip() +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Evolución de los caso por edad y sexo (al: ", last_date, ")") ,
caption = "Fuente: datos.gob.ar",
y = "Casos Confirmados",
x = ""
) +
scale_fill_manual(values = c("firebrick3", "darkorange", "deepskyblue3", "darkorchid1", "seagreen")) +
scale_color_manual(values = rep("Black",5)) +
theme_elegante_std(base_family = "Assistant")
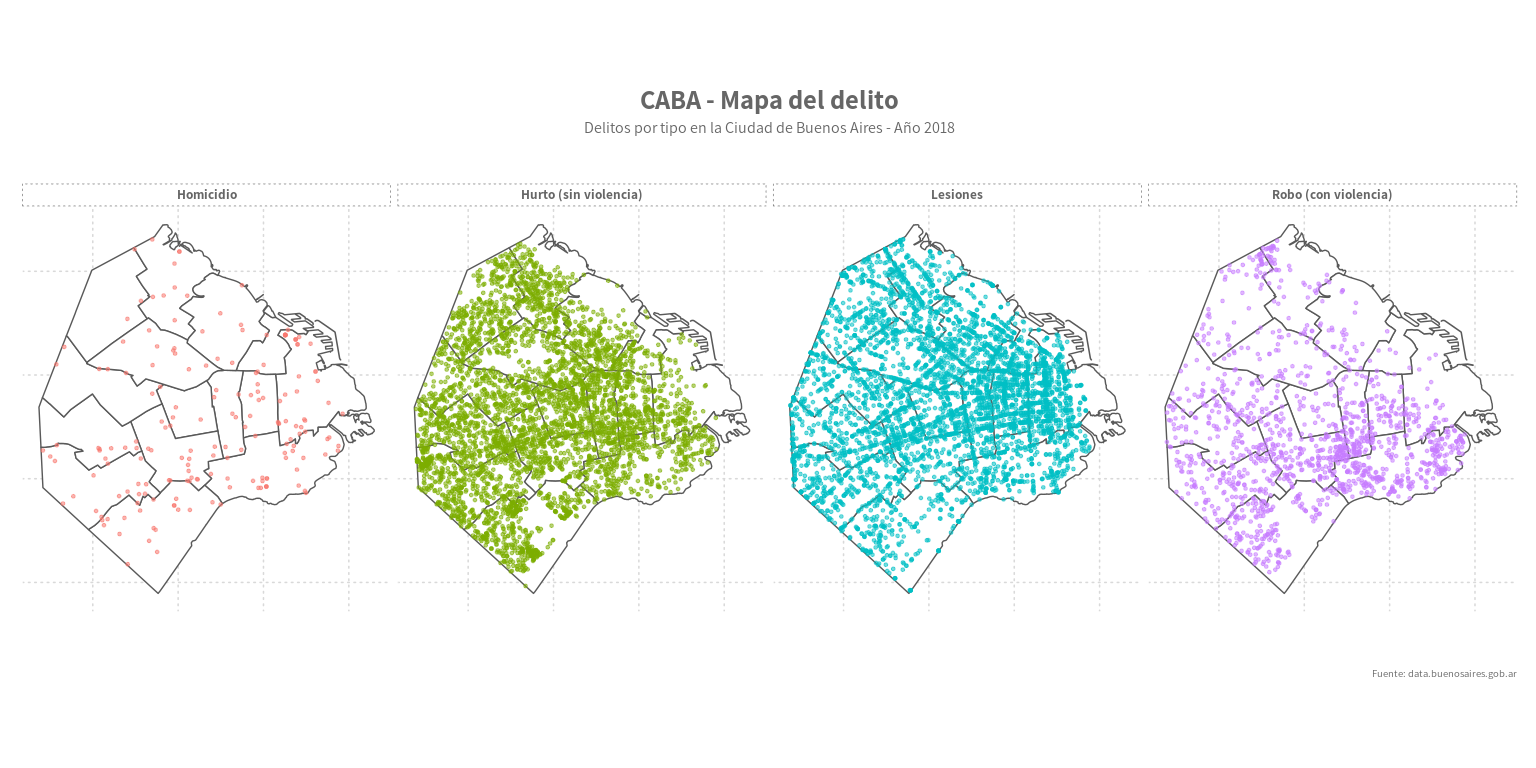
Día 18: Datos espaciales
En este casos hacemos un mapa de puntos (“dot map”) dónde cada punto es una coordenada geográfica y representa una ocurrencia de una variable categórica. Usamos el mapa de la Ciudad de Buenos Aires y la estadísticas de delitos del año 2018, para ubicar estos geográficamente.
library("tidyverse")
library("sf")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# delitos <- read.csv("http://cdn.buenosaires.gob.ar/datosabiertos/datasets/mapa-del-delito/delitos_2019.csv",
# na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# comunas <- st_read('https://bitsandbricks.github.io/data/CABA_comunas.geojson')
delitos <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/delitos.caba.Rda","rb"))
comunas <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/comunas.caba.Rda","rb"))
delitos %>%
na.exclude() %>%
st_as_sf(coords = c("long","lat"), remove = FALSE, crs = 4326) %>%
st_join(comunas)-> delitos_puntos
kable(head(plot_data))
| clasif_edad | sexo | internado | fallecido | n |
|---|---|---|---|---|
| 0 a 6 | Femenino | Ambulatorio | Recuperado | 366 |
| 0 a 6 | Femenino | Internado | Recuperado | 153 |
| 0 a 6 | Masculino | Ambulatorio | Recuperado | 416 |
| 0 a 6 | Masculino | Internado | Recuperado | 165 |
| 7 a 14 | Femenino | Ambulatorio | Recuperado | 552 |
| 7 a 14 | Femenino | Internado | Recuperado | 152 |
La grafica:
ggplot(delitos_puntos) +
geom_sf(data = comunas, fill = "white") +
geom_sf(data = delitos_puntos, aes(color=tipo_delito, alpha = 1), size=1) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("CABA - Mapa del delito"),
subtitle = paste0("Delitos por tipo en la Ciudad de Buenos Aires - Año 2018\n") ,
caption = "Fuente: data.buenosaires.gob.ar",
y = "",
x = ""
) +
theme(axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
legend.position = "none") +
facet_grid(cols = vars(tipo_delito))
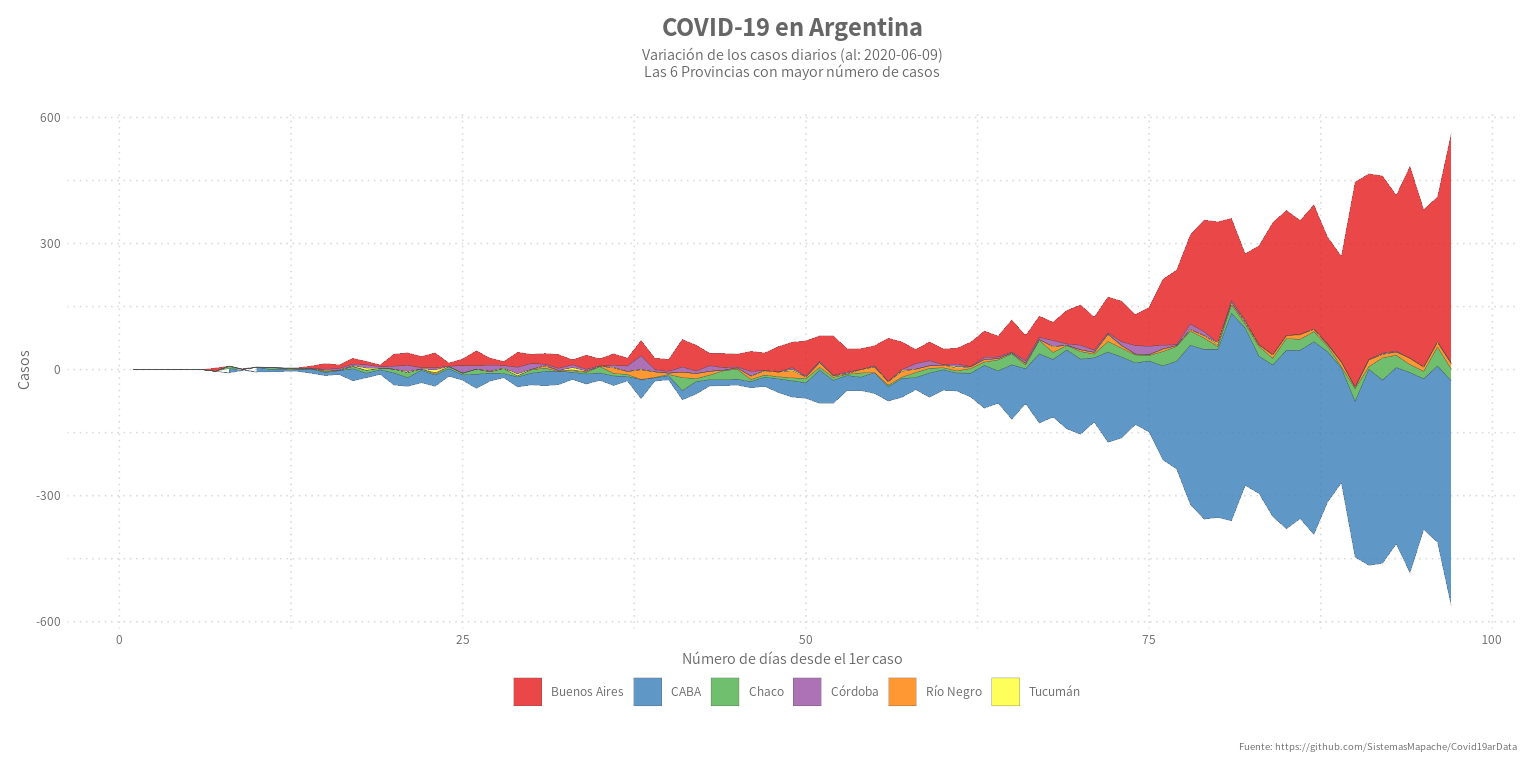
Día 19: Streamgraph
Desde el Catálogo de Visualización de Datos leemos:
También conocido como ThemeRiver.
Este tipo de visualización es una variación del gráfico de área apilada, que en lugar de tener valores en un eje fijo y recto, tiene los valores alrededor de la línea base central variable. Los gráficos de flujo muestran los cambios en los datos a lo largo del tiempo de diferentes categorías a través del uso de formas fluidas y orgánicas que se asemejan un poco a un arroyo. Esto hace que los diagramas de flujo sean estéticamente agradables y más atractivos de mirar.
En un gráfico de flujo, el tamaño de cada forma de flujo individual es proporcional a los valores de cada categoría. El eje que fluye en paralelo al gráfico de flujo se utiliza para dar escala al tiempo. El color puede utilizarse para distinguir cada categoría o para visualizar los valores cuantitativos adicionales en cada categoría a través de la variación del tono de calor.
Los gráficos de flujo son ideales para mostrar conjuntos de datos de alto volumen con el fin de descubrir tendencias y patrones a lo largo del tiempo en una amplia categoría. Por ejemplo, los picos estacionales y los valles en forma de arroyo pueden sugerir un patrón prehistórico. Un diagrama de flujo también puede ser utilizado para un gran grupo de activos durante un cierto período de tiempo.
La desventaja de los diagramas de flujo es que tienen problemas de legibilidad, ya que muy a menudo son confusos en diagramas de flujo con grandes conjuntos de datos. Las categorías con valores más pequeños a menudo se ahogan para dar paso a categorías con valores mucho más grandes, por lo que es imposible ver todos los datos. Además, es imposible leer los valores exactos visualizados, por lo que generalmente se utiliza un código.
Por lo tanto, los diagramas de flujo deben reservarse a audiencias que no tengan intención de pasar mucho tiempo descifrando el gráfico y explorando sus datos. Los diagramas de flujo son mejores para dar una visión más general de los datos. También tienden a funcionar significativamente mejor como una pieza interactiva en lugar de un gráfico estático o impreso.
library("tidyverse")
library("ggTimeSeries")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
first_date <- min(as.Date(covid.data$fecha,"%d/%m/%Y"))
ndias <- max(covid.data$dia_inicio)
dias <- expand.grid(distrito = unique(covid.data$osm_admin_level_4),
dia = 1:ndias,
stringsAsFactors = FALSE)
provincias_de_interes <- c('CABA', 'Buenos Aires', 'Chaco','Córdoba', 'Río Negro', 'Tucumán')
dias %>%
left_join(covid.data, by=c("distrito" = "osm_admin_level_4", "dia" = "dia_inicio")) %>%
mutate(casos = replace_na(nue_casosconf_diff, 0)) %>%
select(dia, distrito, casos) %>%
arrange(distrito, dia) %>%
filter(distrito %in% provincias_de_interes) %>%
ggplot(aes(x = dia, y = casos, group = distrito, fill = distrito)) +
stat_steamgraph(color="gray30", size=.1, alpha=.8) +
theme_elegante_std(base_family = "Assistant") +
scale_fill_brewer(palette="Set1") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Variación de los casos diarios (al: ", last_date, ")\nLas 6 Provincias con mayor número de casos") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Casos",
x = "Número de días desde el 1er caso"
) +
guides(fill = guide_legend(nrow = 1))
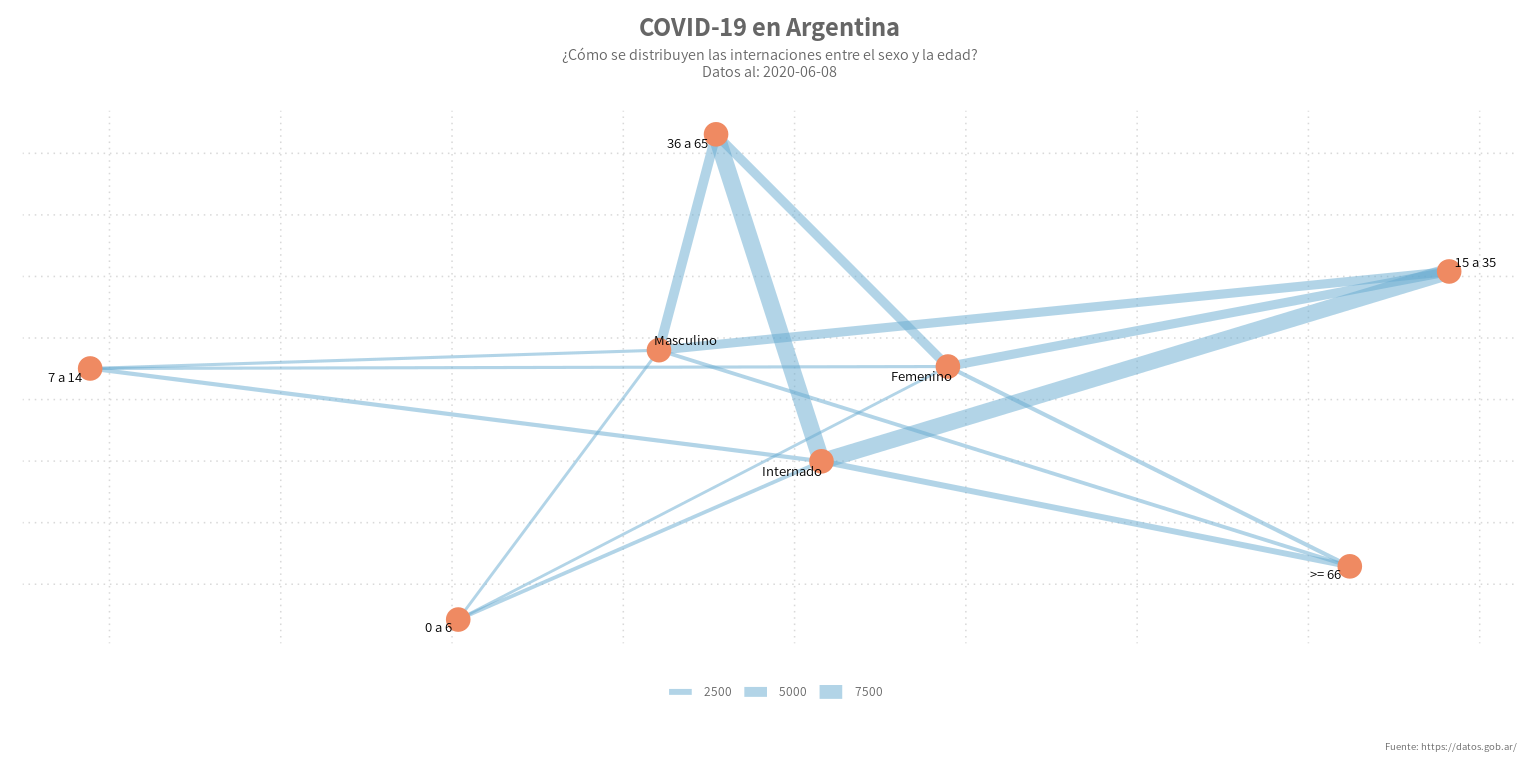
Día 20: Gráfico de redes
library("tidyverse")
library("ggraph")
library("igraph")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read.csv(file='https://sisa.msal.gov.ar/datos/descargas/covid-19/files/Covid19Casos.csv', stringsAsFactors = FALSE, fileEncoding = "UTF-16")
# Datos reproducibles
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.arg.Rda","rb"))
last_date <- max(covid.data$fecha_apertura, na.rm = TRUE)
covid.data %>%
filter(clasificacion_resumen == 'Confirmado',
!is.na(edad),
sexo != 'NR') %>%
mutate(internado = !is.na(fecha_internacion),
cui = !is.na(fecha_cui_intensivo),
arm = replace_na(asistencia_respiratoria_mecanica == "SI",FALSE),
fallecido = replace_na(fallecido == "SI", FALSE),
sexo = ifelse(sexo == 'M', 'Masculino', 'Femenino'),
internado = ifelse(internado, 'Internado', 'Ambulatorio'),
fallecido = ifelse(fallecido, 'Fallecido', 'Recuperado')) %>%
mutate(clasif_edad = case_when(edad <= 6 ~ '0 a 6',
edad > 6 & edad <= 14 ~ '7 a 14',
edad > 14 & edad <= 35 ~ '15 a 35',
edad > 35 & edad <= 65 ~ '36 a 65',
edad > 65 ~ '>= 66')) %>%
select(sexo, edad, clasif_edad, internado, cui, arm, fallecido) %>%
group_by(clasif_edad, sexo, internado, fallecido) %>%
summarise(n = n()) -> data
data %>%
group_by(sexo, clasif_edad) %>%
summarise(n=sum(n)) %>%
select(from=sexo, to=clasif_edad, n) -> sexo_edad
data %>%
group_by(internado, clasif_edad) %>%
summarise(n=sum(n)) %>%
select(to=internado, from=clasif_edad, n) -> internado_edad
internado_edad %>%
rbind(sexo_edad) -> plot_data
kable(head(plot_data))
| to | from | n |
|---|---|---|
| Internado | >= 66 | 2479 |
| Internado | 0 a 6 | 1100 |
| Internado | 15 a 35 | 9003 |
| Internado | 36 a 65 | 9539 |
| Internado | 7 a 14 | 1381 |
| >= 66 | Femenino | 1339 |
La grafica:
plot_data %>%
graph_from_data_frame() %>%
ggraph(layout = 'stress') +
geom_edge_link(aes(edge_width=n), show.legend = TRUE, alpha=0.5, color = "#67a9cf") +
geom_node_point(size=8, color ="#ef8a62") +
geom_node_text(aes(label = name), repel = TRUE, family="Assistant") +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("¿Cómo se distribuyen las internaciones entre el sexo y la edad?\nDatos al: ", last_date) ,
caption = "Fuente: https://datos.gob.ar/",
y = "",
x = ""
) +
guides(fill = guide_legend(nrow = 1)) +
theme(axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank())
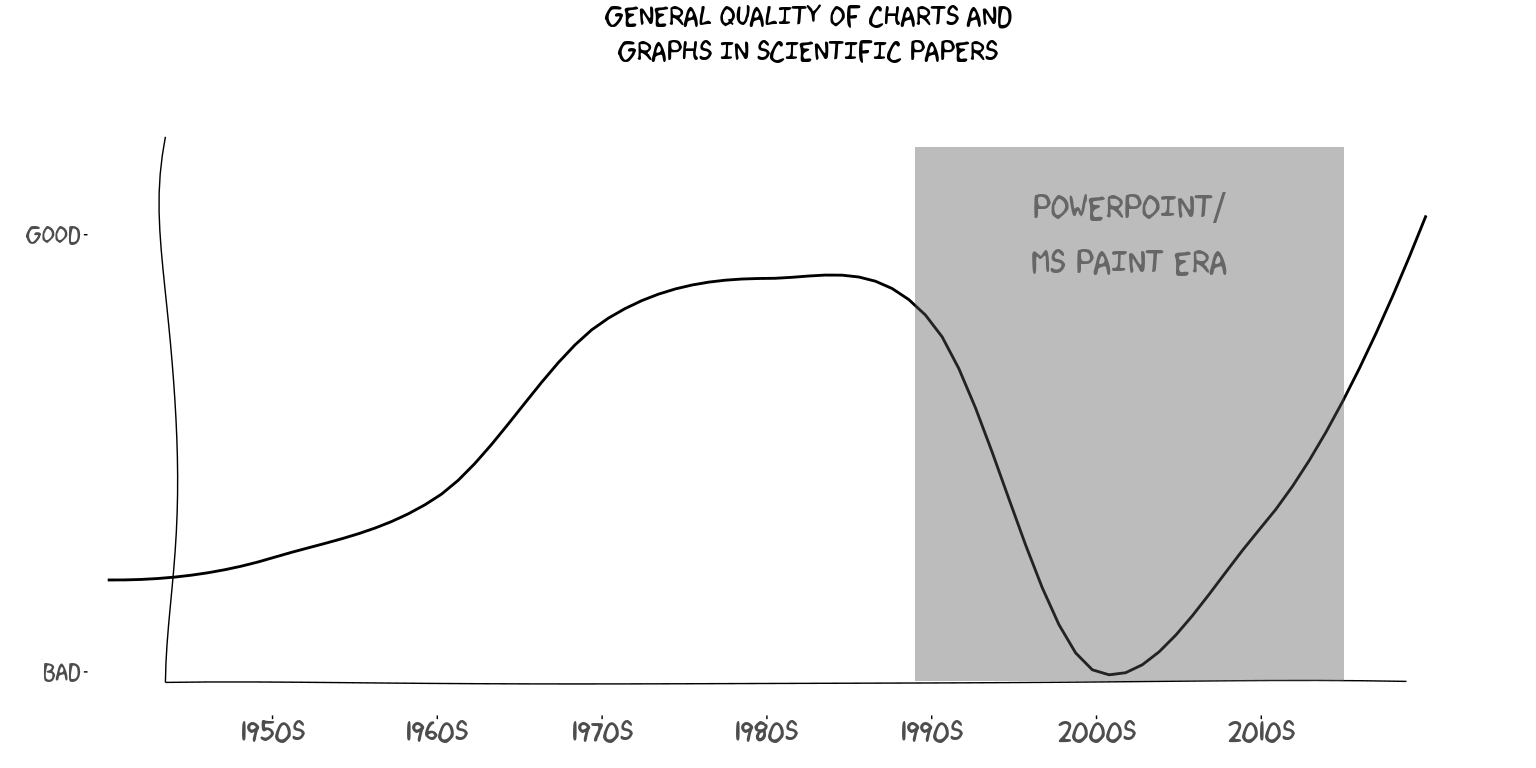
Día 21: Anotaciones
Ejemplo de anotaciones en ggplot, me entretengo un poco armando un clasico “meme”. Para esto necesitamos algunas cosas:
- El Font: [xkcd.ttf]. En linux:
system("mkdir ~/.fonts");system("cp xkcd.ttf ~/.fonts"), luego en R:extrafont::font_import(pattern = "[X/x]kcd", prompt=FALSE);extrafont::loadfonts() - El paquete
xkcd:install.packages("xkcd",dependencies = TRUE)
Usamos [annotate()] que nos permite agregar no solo texto, sino cualquier otro geom.
library("tidyverse")
library("xkcd")
# Para instalar el font xkcd en Linux
# download.file("http://simonsoftware.se/other/xkcd.ttf", dest="xkcd.ttf", mode="wb")
# system("mkdir ~/.fonts")
# system("cp xkcd.ttf ~/.fonts")
# font_import(pattern = "[X/x]kcd", prompt=FALSE)
# loadfonts()
xaxis <- c(1950,1960, 1970, 1980, 1990, 2000, 2010)
xaxislbl <- paste0(xaxis, "s")
data.frame(year=seq(from=1940, to=2020, by=10),
quality=c(1.2, 1.3, 1.4, 1.8, 1.9, 1.8, 1, 1.5, 2)
) -> df
df %>%
ggplot(mapping=aes(x=year, y=quality)) +
geom_smooth(method = 'loess',
color = "black",
formula = 'y ~ x', alpha = 0.2, size = 1, span = .6, se=FALSE) +
labs(title = paste("GENERAL QUALITY OF CHARTS AND\nGRAPHS IN SCIENTIFIC PAPERS\n"),
y = "",
x = ""
) +
xkcdaxis(c(1945,2020),c(1,2.2)) +
annotate("rect", xmin = 1989, xmax = 2015, ymin = .98, ymax = 2.2, alpha = .4) +
annotate("text", x = 2002, y = 2, label = "POWERPOINT/\nMS PAINT ERA", family="xkcd", size=10,) +
scale_y_continuous(breaks = c(1,2), labels = c("BAD", "GOOD")) +
scale_x_continuous(breaks = xaxis, labels = xaxislbl, limits=c(1930, 2020)) +
theme(plot.title=element_text(vjust=1.25, size=24, hjust = 0.5),
axis.text.x = element_text(size=24),
axis.text.y = element_text(size=20)
)
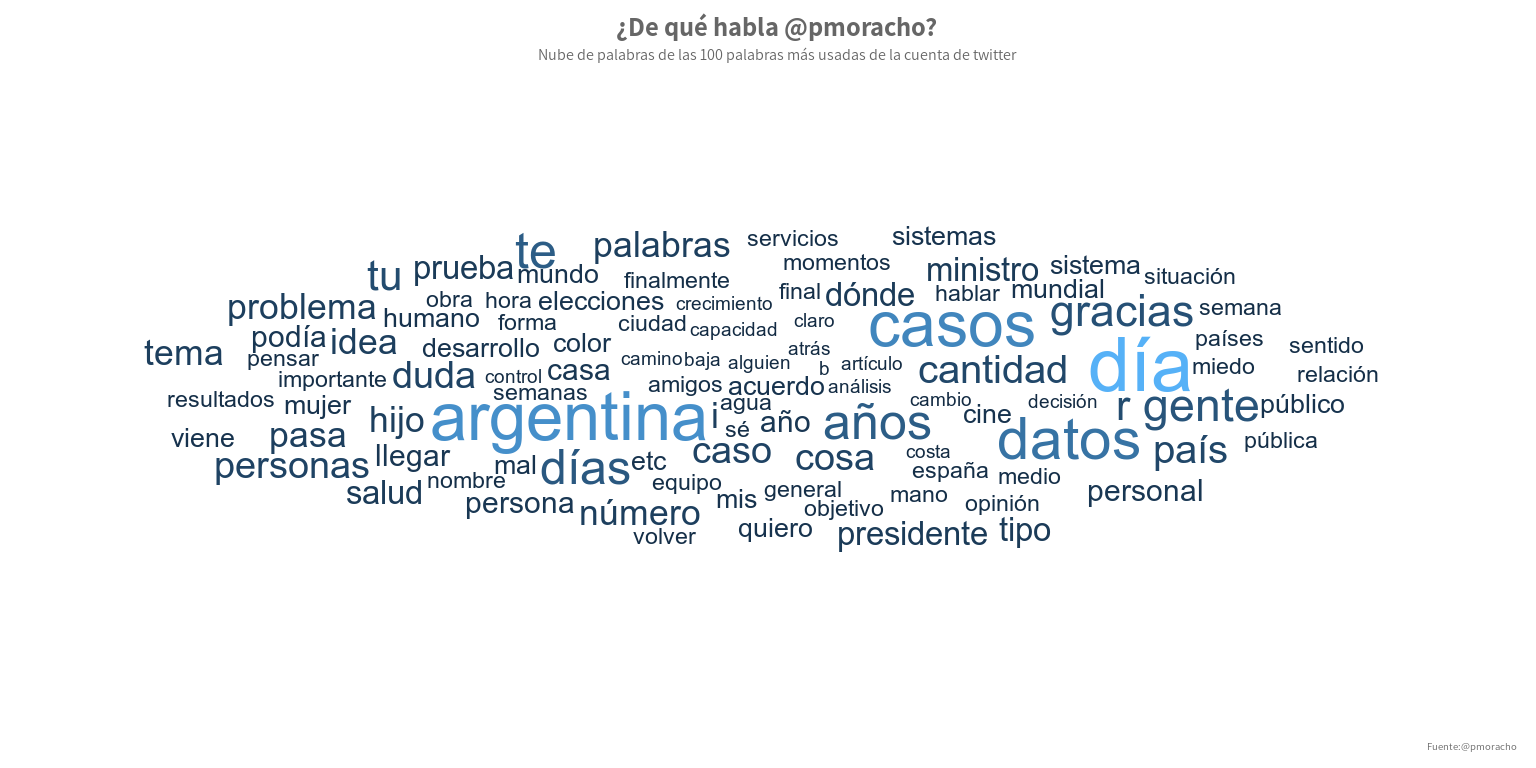
Día 22: visualizar datos textuales
Una clásica “nube de palabras”, en este caso de mi cuenta de twitter. Bastante fácil de implementar con ggwordcloud para la gráfica y rtweet para la parte de extracción de twitter.
library("tidyverse")
library("rtweet")
library("tidytext")
library("ggwordcloud")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# authenticate via web browser
# token <- create_token(
# app = "MarloweApp",
# consumer_key = "",
# consumer_secret = "",
# access_token = "",
# access_secret = "")
# Datos Originales
# RAE_Corpus_1000 <- read.table(file="http://corpus.rae.es/frec/1000_formas.TXT", skip=1, header=FALSE,
# fileEncoding = "Latin1",
# col.names = c("nr", "word", "Frec.absoluta", "Frec.normalizada"),
# stringsAsFactors = FALSE)
#
# stopwords <- read.csv("https://countwordsfree.com/stopwords/spanish/txt",
# col.names="word", header=FALSE, stringsAsFactors = FALSE)
# Datos reproducibles
RAE_Corpus_1000 <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/rae_corpus_1000.Rda","rb"))
stopwords <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/stopwords.Rda","rb"))
twits <- get_timeline("pmoracho", n = 5000)
twits %>%
unnest_tokens(word, text) %>%
select(word) %>%
anti_join(stopwords, by = "word") %>%
inner_join(RAE_Corpus_1000, by = c("word"="word")) -> my_words
my_words %>%
count(word, sort = TRUE) -> my_words_count
my_words %>%
inner_join(my_words_count %>% head(100), by = "word") %>%
# filter(!(word %in% c('https', 'http'))) %>%
count(word) -> plot_data
kable(head(plot_data))
| word | n |
|---|---|
| acuerdo | 4 |
| agua | 3 |
| alguien | 2 |
| amigos | 3 |
| análisis | 2 |
| año | 5 |
La grafica:
plot_data %>%
ggplot(aes(label = word, size=n, color=n)) +
geom_text_wordcloud() +
scale_size_area(max_size = 20) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("¿De qué habla @pmoracho?"),
subtitle = paste0("Nube de palabras de las 100 palabras más usadas de la cuenta de twitter"),
caption = "Fuente:@pmoracho",
y = "",
x = "")
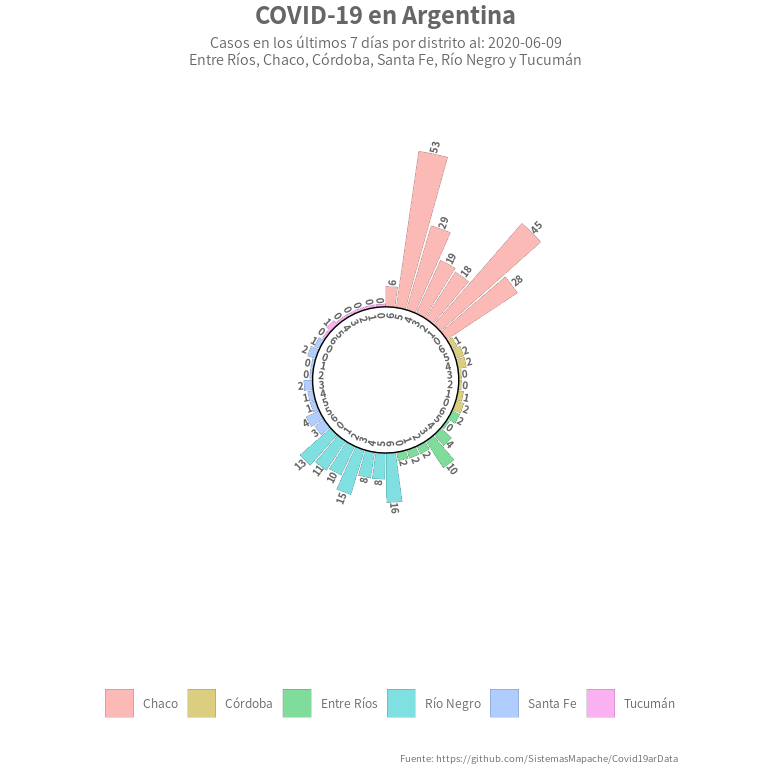
Día 23: Sunburst
library("tidyverse")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
provincias_de_interes <- c('Entre Ríos', 'Chaco', 'Córdoba', 'Santa Fe', 'Río Negro', 'Tucumán')
covid.data %>%
mutate(fecha = as.Date(fecha, "%d/%m/%Y")) %>%
select(dia=dia_inicio, fecha, distrito=osm_admin_level_4, cantidad=nue_casosconf_diff) %>%
filter(dia >= max(dia) - 6,
!(distrito %in% c('Indeterminado'))) %>%
arrange(distrito, dia) %>%
filter(distrito %in% provincias_de_interes) %>%
mutate(cantidad = ifelse(cantidad <= 0, 0, cantidad), nr=row_number()) -> plot_data
plot_data %>%
mutate(angle = 90 - 360 * (nr-0.5) / n(),
hjust = ifelse( angle < -90, 1, 0),
angle = ifelse(angle < -90, angle+180, angle),
text = paste0(cantidad),
text2 = max(dia)-dia) -> label_data
kable(head(plot_data))
| dia | fecha | distrito | cantidad | nr |
|---|---|---|---|---|
| 91 | 2020-06-03 | Chaco | 6 | 1 |
| 92 | 2020-06-04 | Chaco | 53 | 2 |
| 93 | 2020-06-05 | Chaco | 29 | 3 |
| 94 | 2020-06-06 | Chaco | 19 | 4 |
| 95 | 2020-06-07 | Chaco | 18 | 5 |
| 96 | 2020-06-08 | Chaco | 45 | 6 |
kable(head(label_data))
| dia | fecha | distrito | cantidad | nr | angle | hjust | text | text2 |
|---|---|---|---|---|---|---|---|---|
| 91 | 2020-06-03 | Chaco | 6 | 1 | 85.90909 | 0 | 6 | 6 |
| 92 | 2020-06-04 | Chaco | 53 | 2 | 77.72727 | 0 | 53 | 5 |
| 93 | 2020-06-05 | Chaco | 29 | 3 | 69.54545 | 0 | 29 | 4 |
| 94 | 2020-06-06 | Chaco | 19 | 4 | 61.36364 | 0 | 19 | 3 |
| 95 | 2020-06-07 | Chaco | 18 | 5 | 53.18182 | 0 | 18 | 2 |
| 96 | 2020-06-08 | Chaco | 45 | 6 | 45.00000 | 0 | 45 | 1 |
La grafica:
plot_data %>%
ggplot(aes(x=nr, y=cantidad+1, fill=distrito)) +
geom_bar(stat="identity", alpha=0.5, color="black", size=.05) +
ylim(-25,max(plot_data$cantidad)+2) +
geom_hline(aes(yintercept=0)) +
geom_text(data=label_data, aes(x=nr, y=cantidad+1.2, label=text, hjust=hjust), color="black", fontface="bold",alpha=0.6, size=3, angle = label_data$angle, inherit.aes = FALSE ) +
geom_text(data=label_data, aes(x=nr, y=-4, label=text2, hjust=hjust), color="black", fontface="bold",alpha=0.6, size=3, angle = label_data$angle, inherit.aes = FALSE ) +
coord_polar() +
theme_elegante_std(base_family = "Assistant") +
labs(title = "COVID-19 en Argentina",
subtitle = paste0("Casos en los últimos 7 días por distrito al: ", last_date, "\nEntre Ríos, Chaco, Córdoba, Santa Fe, Río Negro y Tucumán"),
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "",
x = "") +
guides(fill = guide_legend(nrow = 1)) +
theme(
legend.position = "bottom",
axis.line.x = element_blank(),
axis.line.y = element_blank(),
axis.line= element_blank(),
axis.text = element_blank(),
axis.title = element_blank(),
axis.title.x = element_blank(),
axis.text.x = element_blank(),
axis.ticks.x = element_blank(),
axis.title.y = element_blank(),
axis.text.y = element_blank(),
axis.ticks.y = element_blank(),
panel.grid = element_blank(),
plot.margin = unit(rep(.2,4), "cm"),
axis.ticks = element_blank(),
legend.background = element_blank(),
legend.key = element_blank(),
panel.background = element_blank(),
panel.border = element_blank(),
strip.background = element_blank(),
plot.background = element_blank(),
panel.grid.major = element_blank(),
panel.grid.minor = element_blank()
)
Día 24: Coropletas
library("tidyverse")
library("sf")
library("viridis")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# delitos <- read.csv("http://cdn.buenosaires.gob.ar/datosabiertos/datasets/mapa-del-delito/delitos_2019.csv",
# na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# comunas <- st_read('https://bitsandbricks.github.io/data/CABA_comunas.geojson')
delitos <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/delitos.caba.Rda","rb"))
comunas <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/comunas.caba.Rda","rb"))
delitos %>%
na.exclude() %>%
st_as_sf(coords = c("long","lat"), remove = FALSE, crs = 4326) %>%
st_join(comunas)-> delitos_puntos
delitos_puntos %>%
mutate(n_delito = match(tipo_delito, c("Lesiones", "Hurto (sin violencia)","Robo (con violencia)","Homicidio")),
comunas = factor(comuna)) %>%
group_by(barrios,comunas) %>%
summarize(nrank = n()) %>%
as.data.frame() %>%
select(barrios, comunas, nrank) -> ranking_comunas
comunas %>%
left_join(ranking_comunas, by = "comunas") -> plot_data
kable(head(plot_data))
| barrios.x | perimetro | area | comunas | barrios.y | nrank | geometry |
|---|---|---|---|---|---|---|
| CONSTITUCION - MONSERRAT - PUERTO MADERO - RETIRO - SAN NICOLAS - SAN TELMO | 35572.65 | 17802807 | 1 | CONSTITUCION - MONSERRAT - PUERTO MADERO - RETIRO - SAN NICOLAS - SAN TELMO | 814 | MULTIPOLYGON (((-58.36854 -… |
| RECOLETA | 21246.61 | 6140873 | 2 | RECOLETA | 296 | MULTIPOLYGON (((-58.39521 -… |
| BALVANERA - SAN CRISTOBAL | 10486.26 | 6385991 | 3 | BALVANERA - SAN CRISTOBAL | 651 | MULTIPOLYGON (((-58.41192 -… |
| BARRACAS - BOCA - NUEVA POMPEYA - PARQUE PATRICIOS | 36277.44 | 21701236 | 4 | BARRACAS - BOCA - NUEVA POMPEYA - PARQUE PATRICIOS | 1069 | MULTIPOLYGON (((-58.3552 -3… |
| ALMAGRO - BOEDO | 12323.47 | 6660526 | 5 | ALMAGRO - BOEDO | 643 | MULTIPOLYGON (((-58.41287 -… |
| CABALLITO | 10990.96 | 6851029 | 6 | CABALLITO | 536 | MULTIPOLYGON (((-58.43061 -… |
La grafica:
plot_data %>%
ggplot() +
geom_sf(aes(fill = nrank), color="gray60") +
scale_fill_viridis(option = "inferno", direction = -1) +
theme_elegante_std(base_family = "Assistant") +
labs(title = paste("CABA - Mapa del delito"),
subtitle = paste0("Nivel de peligrosidad por comuna - Año 2018\n") ,
caption = "Fuente: data.buenosaires.gob.ar",
y = "",
x = "",
fill = "Delitos"
) +
theme(axis.text.x=element_blank(),
axis.ticks.x=element_blank(),
axis.title.y=element_blank(),
axis.text.y=element_blank(),
axis.ticks.y=element_blank(),
legend.title = element_text(face="bold"),
legend.position = "right")
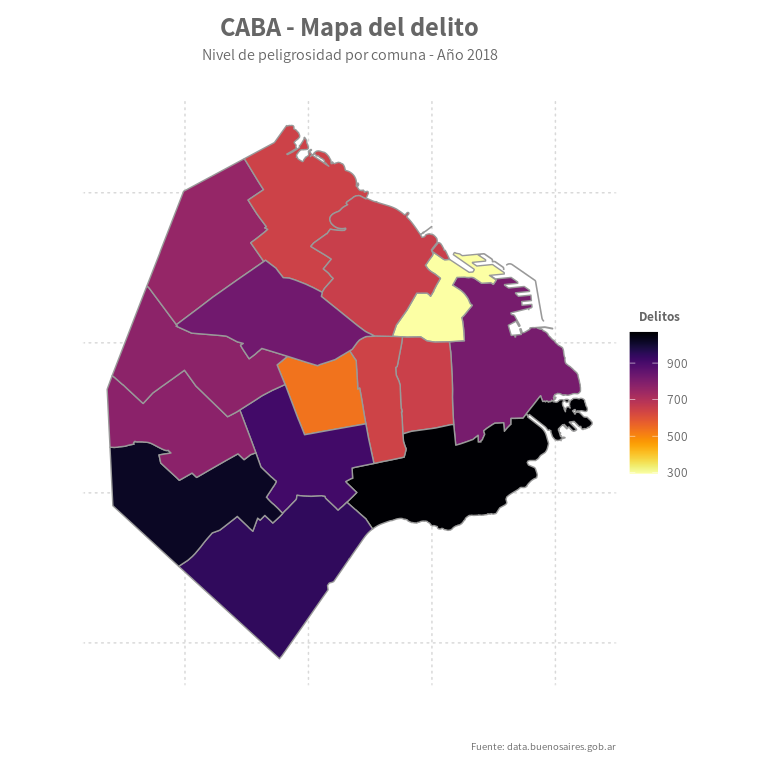
Día 25: Gráficas de violín
Los datos:
library("tidyverse")
library("ggrepel")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
covid.data %>%
filter(osm_admin_level_4 %in% c('CABA', 'Buenos Aires')) %>%
mutate(distrito = factor(osm_admin_level_4)) %>%
select(dia=dia_inicio, distrito, cantidad=nue_casosconf_diff) -> plot_data
kable(head(plot_data))
| dia | distrito | cantidad |
|---|---|---|
| 1 | CABA | 1 |
| 4 | Buenos Aires | 1 |
| 7 | Buenos Aires | 8 |
| 8 | CABA | 1 |
| 9 | Buenos Aires | 1 |
| 9 | CABA | 1 |
El gráfico:
plot_data %>%
ggplot(aes(x=distrito, y=cantidad, fill=distrito)) +
geom_violin() +
geom_boxplot(color="gray50", alpha = 0.5, width=0.05) +
# scale_y_log10() +
# stat_summary(fun=mean, geom="point", size=2, color = "blue") +
stat_summary(fun=median, geom="point", size=2) +
geom_text_repel(data = plot_data %>%
group_by(distrito) %>%
summarise(mediana = median(cantidad)),
aes(x = distrito, y = mediana, label=paste("Mediana:",mediana)),
family = "Ralleway",
vjust= 8,
hjust= -2) +
geom_text_repel(data = plot_data %>%
group_by(distrito) %>%
summarise(maximo = max(cantidad)),
aes(x = distrito, y = maximo, label=paste("Cantidad máxima en un día:",maximo, "casos")),
family = "Ralleway",
vjust= -8,
hjust= 2) +
coord_flip() +
scale_fill_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Ralleway") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste("Distribución de los casos diarios en CABA y Buenos Aires al: ", last_date) ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "Cantidad de casos diarios",
x = ""
) +
theme(legend.position = "none")
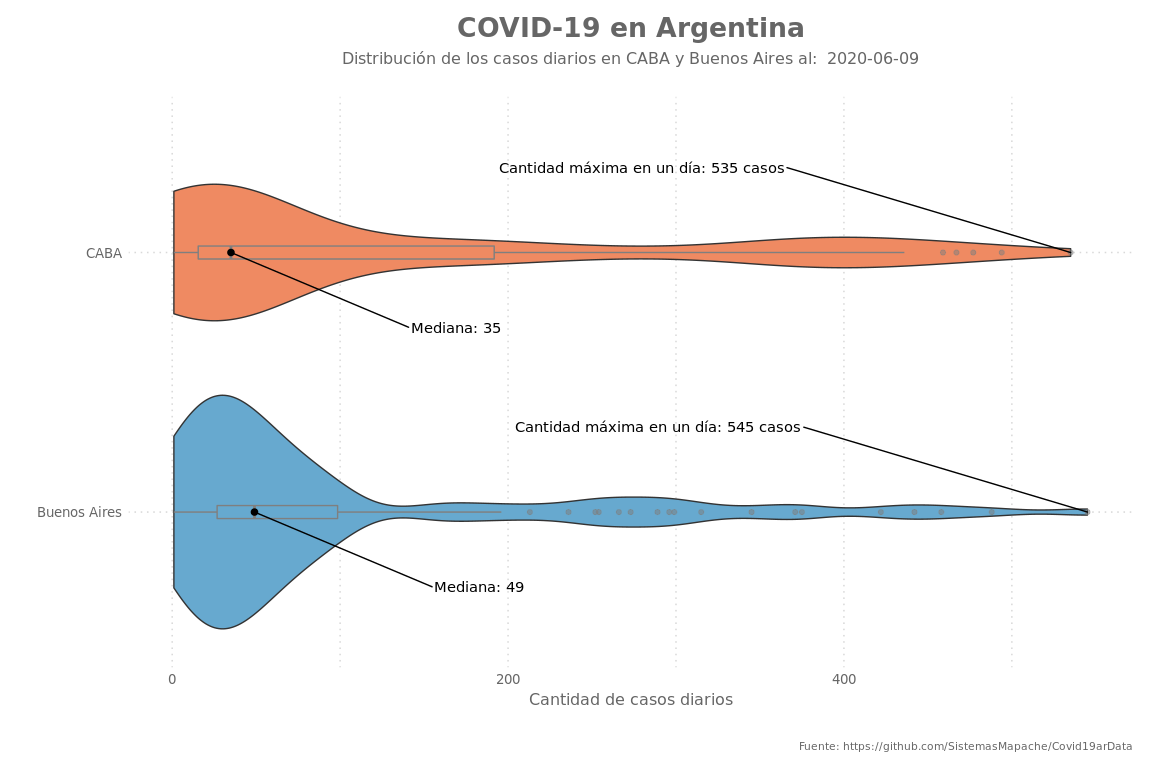
Día 26: Gráfica de Marimeko
Los datos:
library("tidyverse")
library("ggmosaic") # devtools::install_github("haleyjeppson/ggmosaic")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# delitos <- read.csv("http://cdn.buenosaires.gob.ar/datosabiertos/datasets/mapa-del-delito/delitos_2019.csv",
# na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
delitos <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/delitos.caba.Rda","rb"))
delitos %>%
mutate(tipo_delito=factor(tipo_delito, levels = c("Lesiones", "Hurto (sin violencia)", "Robo (con violencia)", "Homicidio")),
comuna = factor(comuna)) %>%
na.exclude() %>%
select(comuna, tipo_delito) -> plot_data
kable(head(plot_data))
| comuna | tipo_delito | |
|---|---|---|
| 1 | 4 | Lesiones |
| 3 | 15 | Lesiones |
| 4 | 10 | Hurto (sin violencia) |
| 5 | 4 | Robo (con violencia) |
| 7 | 9 | Lesiones |
| 8 | 12 | Hurto (sin violencia) |
El gráfico:
plot_data %>%
ggplot() +
geom_mosaic(aes(x = product(comuna), fill=tipo_delito), offset= 0.003) +
theme_elegante_std(base_family = "Ralleway") +
labs(title = "Delitos en CABA",
subtitle = paste0("Por Comuna (año 2018)"),
caption = "Fuente: data.buenosaires.gob.ar",
y = "",
x = "Comunas"
) +
scale_fill_manual(values=c("#56B4E9", "#009E73", "#0072B2", "#E69F00"))
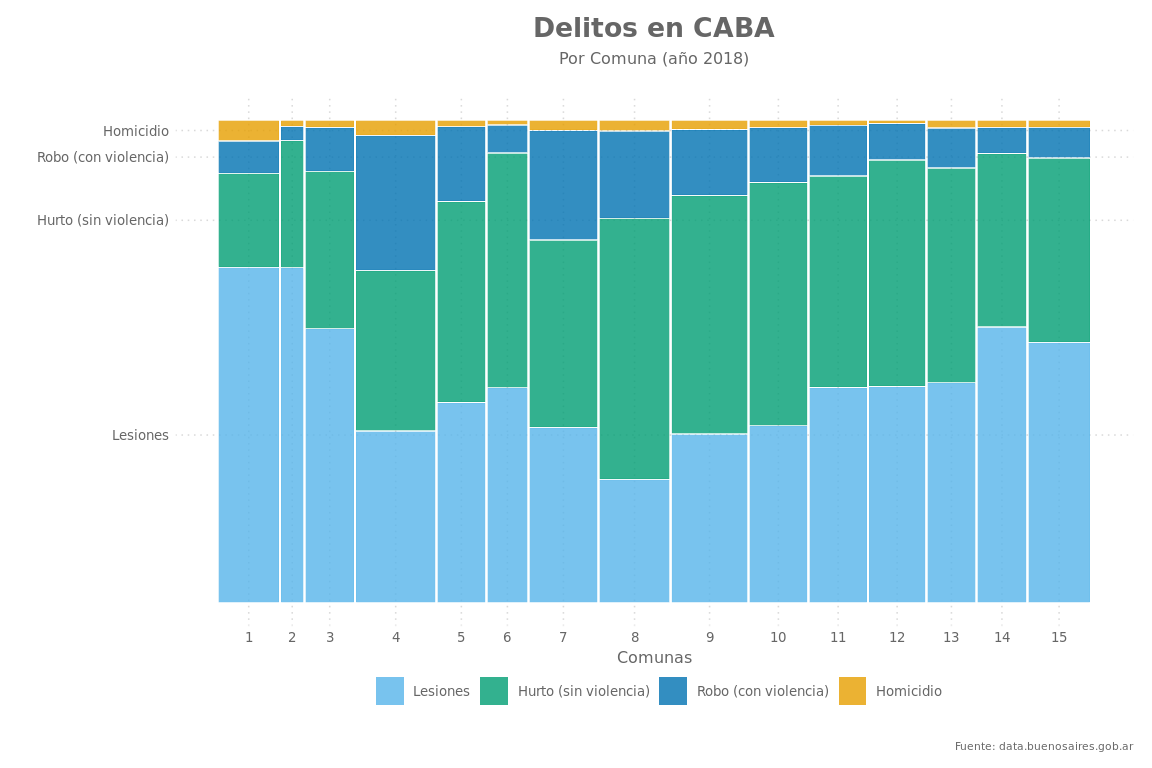
Día 27: Gráficos animados
Los gráficos animados son una muy útil herramienta para visualizar la evolución de variables, por lo general en el tiempo. En este ejemplo, transformo una de las anteriores gráficas (lollipop) en una gráfica animada.
Los datos:
library("tidyverse")
library("countrycode")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Para descarga de los datos actualizados
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM", stringsAsFactors = FALSE)
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
rango_fechas <- range(unique(as.Date(covid.data$dateRep,"%d/%m/%Y")))
todas_las_fecha <- seq(from=rango_fechas[1], to=rango_fechas[2], by=1)
todos_los_paises <- unique(covid.data$countryterritoryCode)
todos_los_paises <- todos_los_paises[!is.na(todos_los_paises)]
expand_grid(countryterritoryCode=todos_los_paises,
fecha=todas_las_fecha) %>%
left_join(covid.data %>%
mutate(fecha = as.Date(dateRep, "%d/%m/%Y")),
by=c("countryterritoryCode", "fecha")) %>%
inner_join(codelist, by = c("countryterritoryCode" = "iso3c")) %>%
mutate(pais = ifelse(is.na(un.name.es), iso.name.en, un.name.es),
continente = continent) %>%
group_by(pais, continente) %>%
mutate(casos = cumsum(replace_na(cases, 0)),
fallecidos = cumsum(replace_na(deaths, 0))) %>%
select(pais, continente, fecha, casos, fallecidos) %>%
mutate(dia=row_number()) %>%
ungroup() %>%
select(pais, dia, fecha, casos) %>%
group_by(fecha) %>%
arrange(-casos) %>%
mutate(nr = row_number(),
media = mean(casos),
sobre_media = casos > media) %>%
select(pais, fecha, casos, media, sobre_media, nr) %>%
filter(nr <= 50,
fecha >= '2020-02-01') %>%
arrange(fecha, nr) -> plot_data
out_folder <- tempdir(check = TRUE)
fechas <- unique(plot_data$fecha)
kable(head(plot_data))
| pais | fecha | casos | media | sobre_media | nr |
|---|---|---|---|---|---|
| China | 2020-02-01 | 11809 | 58.79803 | TRUE | 1 |
| Tailandia | 2020-02-01 | 19 | 58.79803 | FALSE | 2 |
| Singapur | 2020-02-01 | 16 | 58.79803 | FALSE | 3 |
| Japón | 2020-02-01 | 15 | 58.79803 | FALSE | 4 |
| República de Corea | 2020-02-01 | 12 | 58.79803 | FALSE | 5 |
| Australia | 2020-02-01 | 9 | 58.79803 | FALSE | 6 |
La gráfica
for (f in 1:length(fechas)) {
fecha_actual = fechas[f]
max_casos = max(plot_data$casos[plot_data$fecha == fecha_actual])
plot_data %>%
filter(fecha == fecha_actual) %>%
ggplot(aes(x=fct_reorder(pais, -nr), y = casos, color = sobre_media)) +
geom_segment(aes( y=0 , xend = pais, yend = casos)) +
geom_point() +
coord_flip() +
geom_text(aes(label = casos,
vjust = .5,
hjust = -.1),
position = position_dodge(width=1)) +
labs(title = paste("COVID-19 en el mundo"),
subtitle = paste("Casos por pais al: ", fecha_actual, '\nLos primeros 50 países') ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "Casos",
x = ""
) +
scale_y_continuous(breaks = scales::pretty_breaks(n = 5), limits = c(0, max_casos * 1.03),
labels = scales::comma) +
scale_color_manual(labels = c("Sobre la media", "Debajo de la media"), values = c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Ralleway") -> p
ggsave(filename = paste0(out_folder, "/", str_pad(f, 6, pad = "0"), ".png"), plot =p, width = 36, height = 22, units = "cm", dpi = 100)
}
system(paste0('ffmpeg -y -framerate 2 -i ', out_folder, '/%06d.png -c:v libx264 -r 30 ', getwd(), '/dia27.mp4'))
system(paste0('ffmpeg -y -ss 30 -t 3 -i ', getwd(), '/dia27.mp4', '-vf "fps=10,scale=1028:-1:flags=lanczos,split[s0][s1];[s0]palettegen[p];[s1][p]paletteuse" -loop 0'), getwd(), '/dia27.gif')
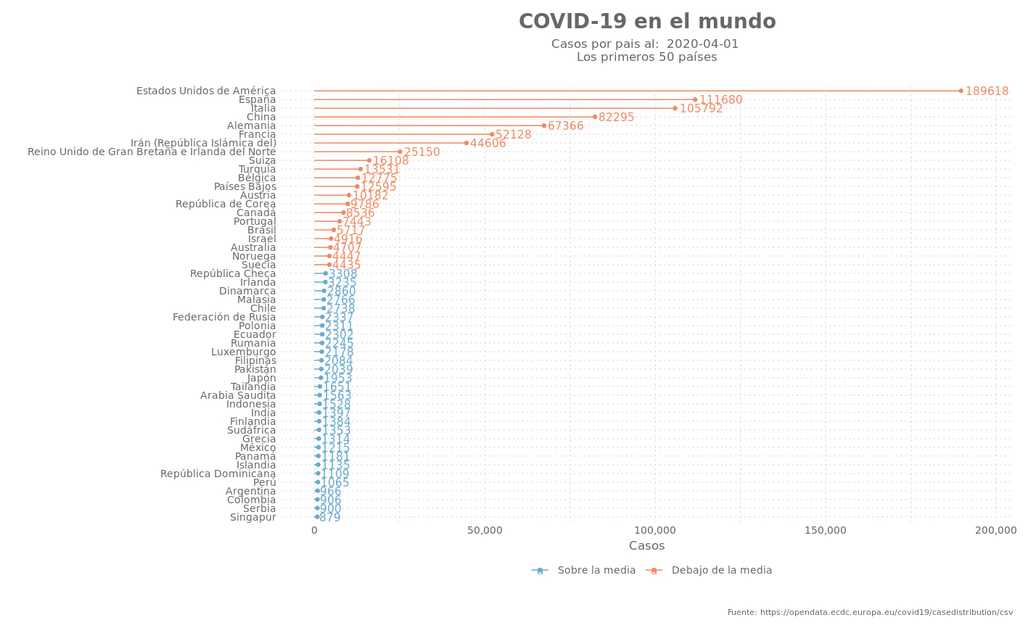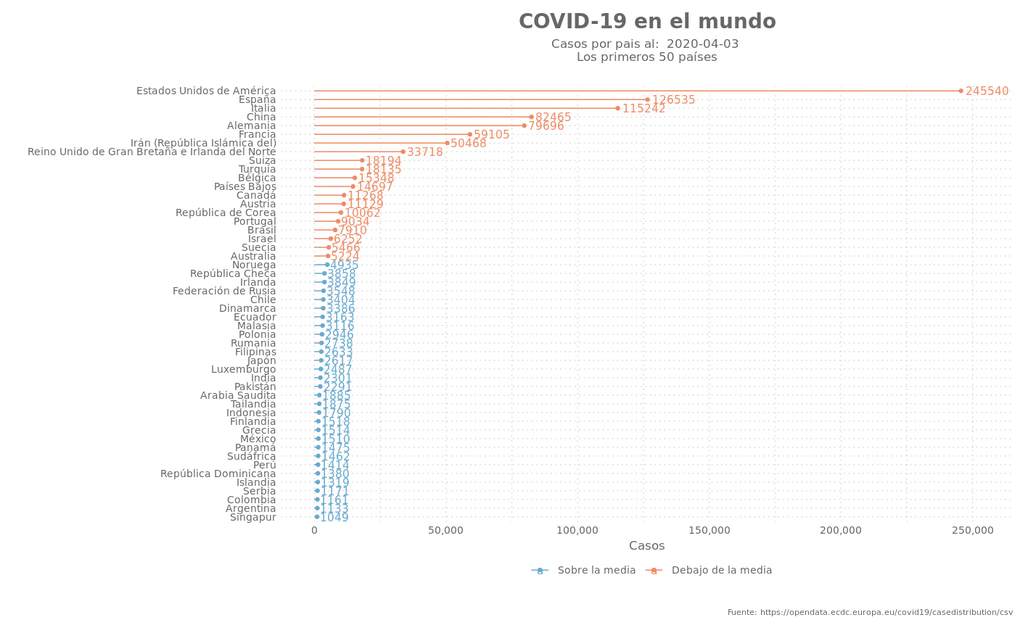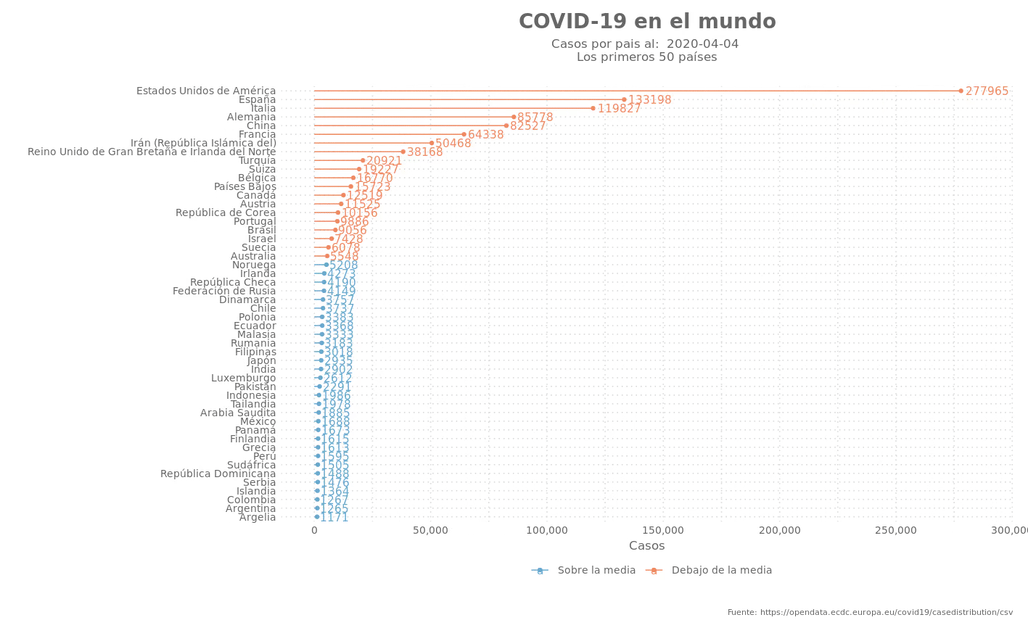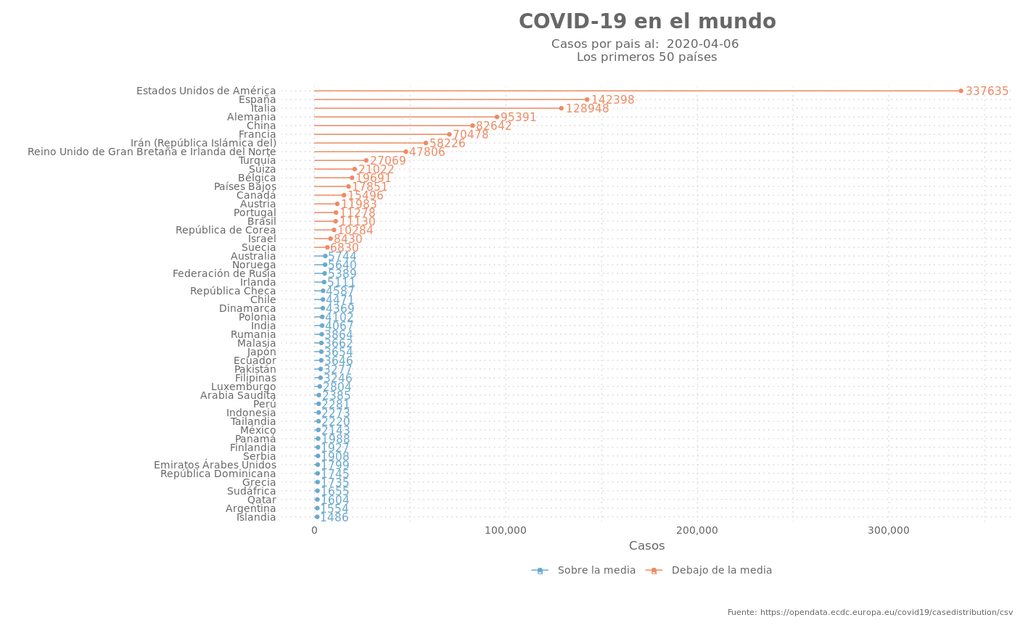
Día 28: Diagramas de cuerdas
Los diagramas de cuerda se usan para mostrar relaciones entre entidades (variables categoricas), la importancia de esa relación puede estar definido por una variable continua. De alguna manera es una variante de los diagramas de redes, pero organizado de forma circular. Este tipo de diagramas se popularizado en 2007 gracias a su uso en las infografías presentadas por el New York Times sobre el genoma humano.
NOTA: Con este lamentablemente se murió la esperanza de resolver todo gon ggplot2
Los datos:
library("circlize")
library("tidyverse")
# Datos originales
# covid.data <- read.csv(file='https://sisa.msal.gov.ar/datos/descargas/covid-19/files/Covid19Casos.csv', stringsAsFactors = FALSE, fileEncoding = "UTF-16")
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.arg.Rda","rb"))
last_date <- max(covid.data$fecha_apertura, na.rm = TRUE)
covid.data %>%
filter(clasificacion_resumen == 'Confirmado',
!is.na(edad),
sexo != 'NR') %>%
mutate(internado = !is.na(fecha_internacion),
cui = !is.na(fecha_cui_intensivo),
arm = replace_na(asistencia_respiratoria_mecanica == "SI",FALSE),
fallecido = replace_na(fallecido == "SI", FALSE),
sexo = ifelse(sexo == 'M', 'Masculino', 'Femenino'),
internado = ifelse(fecha_internacion != "", 'Internado', 'Ambulatorio'),
fallecido = ifelse(fallecido == "SI", 'Fallecido', 'Recuperado')) %>%
mutate(clasif_edad = case_when(edad <= 6 ~ '0 a 6',
edad > 6 & edad <= 14 ~ '7 a 14',
edad > 14 & edad <= 35 ~ '15 a 35',
edad > 35 & edad <= 65 ~ '36 a 65',
edad > 65 ~ '>= 66')) %>%
select(sexo, edad, clasif_edad, internado, cui, arm, fallecido) %>%
mutate(clasif_edad = factor(clasif_edad, c('0 a 6', '7 a 14', '15 a 35', '36 a 65', '>= 66')),
internado = factor(internado, c("Ambulatorio", "Internado")),
fallecido = factor(fallecido, c("Recuperado", "Fallecido"))
) %>%
group_by(clasif_edad, sexo, internado, fallecido) %>%
summarise(n = n()) -> plot_data
kable(head(plot_data))
| clasif_edad | sexo | internado | fallecido | n |
|---|---|---|---|---|
| 0 a 6 | Femenino | Ambulatorio | Recuperado | 366 |
| 0 a 6 | Femenino | Internado | Recuperado | 153 |
| 0 a 6 | Masculino | Ambulatorio | Recuperado | 416 |
| 0 a 6 | Masculino | Internado | Recuperado | 165 |
| 7 a 14 | Femenino | Ambulatorio | Recuperado | 552 |
| 7 a 14 | Femenino | Internado | Recuperado | 153 |
La gráfica:
plot_data %>%
group_by(clasif_edad, sexo) %>%
summarize(n = sum(n)) %>%
select(from=clasif_edad, to=sexo, value=n) %>%
chordDiagram()
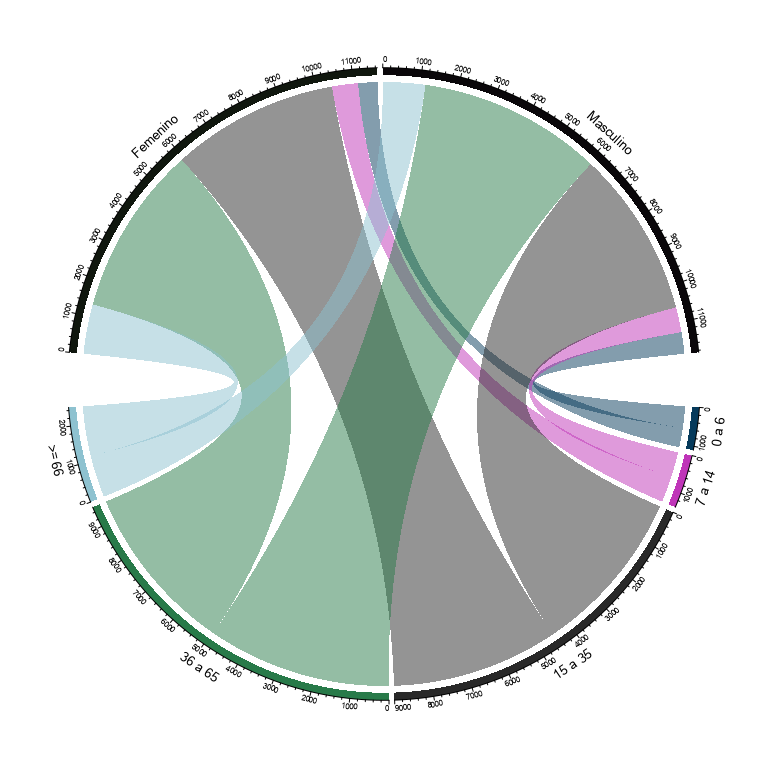
Día 29: gráficos de coordenadas paralelas
Los datos:
library("tidyverse")
library("GGally")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read.csv("https://opendata.ecdc.europa.eu/covid19/casedistribution/csv", na.strings = "", fileEncoding = "UTF-8-BOM",
# stringsAsFactors = FALSE)
# Datos para reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.mundial.Rda","rb"))
covid.data %>%
mutate(fecha = as.Date(dateRep, '%d/%m/%Y'),
pais = countriesAndTerritories) %>%
filter(continentExp %in% c('America')) %>%
# filter(pais %in% c('Argentina', 'Brazil', 'Chile', 'Paraguay', 'Uruguay', 'Colombia', 'Bolivia', 'Ecuador', 'Peru', 'Venezuela', 'México')) %>%
group_by(pais, popData2019) %>%
summarise(casos = sum(cases), fallecidos = sum(deaths)) %>%
arrange(-casos) %>%
head(10) -> plot_data
kable(head(plot_data))
| pais | popData2019 | casos | fallecidos |
|---|---|---|---|
| United_States_of_America | 329064917 | 2191052 | 118434 |
| Brazil | 211049519 | 978142 | 47748 |
| Peru | 32510462 | 244388 | 7461 |
| Chile | 18952035 | 225103 | 3841 |
| Mexico | 127575529 | 165455 | 19747 |
| Canada | 37411038 | 100209 | 8300 |
La gráfica:
plot_data %>%
ggparcoord(columns = 2:4, groupColumn = 1) +
theme_elegante_std(base_family = "Ralleway") +
scale_x_discrete(labels = c("Población", "Casos", "Fallecidos")) +
labs(title = paste("COVID-19"),
subtitle = paste("Relación Población / Casos / fallecidos (Top 10 América)") ,
caption = "Fuente: https://opendata.ecdc.europa.eu/covid19/casedistribution/csv",
y = "",
x = ""
)
Día 30: diagramas de área polar o de Florence Nightingale
Los datos:
library("tidyverse")
if ("ggelegant" %in% rownames(installed.packages())) {
library("ggelegant")
} else {
# devtools::install_github("pmoracho/ggelegant")
theme_elegante_std <- function(base_family) {}
}
# Datos originales
# covid.data <- read_csv('https://docs.google.com/spreadsheets/d/16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA/export?format=csv&id=16-bnsDdmmgtSxdWbVMboIHo5FRuz76DBxsz_BbsEVWA&gid=0')
# Datos reproducir la gráfica
covid.data <- readRDS(url("https://github.com/pmoracho/R/raw/master/data/covid.casos.arg.Rda","rb"))
last_date <- max(as.Date(covid.data$fecha,"%d/%m/%Y"))
covid.data %>%
filter(osm_admin_level_4 %in% c('CABA', 'Buenos Aires', 'Chaco', 'Río Negro', 'Córdoba')) %>%
group_by(distrito = osm_admin_level_4) %>%
summarise(casos = sum(nue_casosconf_diff), fallecidos=sum(nue_fallecidos_diff)) %>%
pivot_longer(-distrito) -> plot_data
kable(head(plot_data))
| distrito | name | value |
|---|---|---|
| Buenos Aires | casos | 9590 |
| Buenos Aires | fallecidos | 304 |
| CABA | casos | 11965 |
| CABA | fallecidos | 262 |
| Chaco | casos | 1118 |
| Chaco | fallecidos | 64 |
La gráfica:
plot_data %>%
ggplot(aes(x = distrito, y=value, fill = name)) +
geom_col(width = 1, color = "black") +
scale_y_sqrt() +
coord_polar(start=3*pi/2) +
scale_fill_discrete(palette = function(x) c("#67a9cf", "#ef8a62")) +
theme_elegante_std(base_family = "Ralleway") +
labs(title = paste("COVID-19 en Argentina"),
subtitle = paste0("Total de casos y fallecidos por distrito (al: ", last_date, ")") ,
caption = "Fuente: https://github.com/SistemasMapache/Covid19arData",
y = "",
x = ""
) +
theme(axis.text.y = element_blank())